- Home /
- Publicaciones de patentes /
- MARCADORES DE EFICIENCIA METABÓLICA
Patente nacional por "MARCADORES DE EFICIENCIA METABÓLICA"
Este registro ha sido solicitado por
FUNDACIÓN UNIVERSIDAD FRANCISCO DE VITORIA
a través del representanteGUSTAVO ADOLFO DE DIOS SERRANÍA
Contacto
- Estado: Vigente
- País:
- España
- Fecha solicitud:
- 15/07/2022
- Número solicitud:
-
P202230654
- Número publicación:
-
ES2958596
- Fecha de concesión:
-
- Inventores:
-
Persona física
- Datos del titular:
-
Fundación Universidad Francisco de Vitoria
- Datos del representante:
-
Gustavo Adolfo de Dios Serranía
- Clasificación Internacional de Patentes:
- C12Q 1/68
- Clasificación Internacional de Patentes de la publicación:
- C12Q 1/68
- Fecha de vencimiento:
Quiero registrar una patente
Reivindicaciones:
+ ES-2958596_A11.- Método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1-1+, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) o, - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) es indicativo de eficiencia metabólica. 2. Método según la reivindicación 1, donde la eficiencia metabólica es indicativa de talento deportivo. 3. Método según la reivindicación 2, donde el talento deportivo está asociado a deportes de resistencia. 4. Método según cualquiera de las reivindicaciones anteriores, donde la muestra es una muestra de sangre, mucosa bucal o saliva. 5. Método según cualquiera de las reivindicaciones anteriores donde la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. 6. Uso de los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- como marcadores de eficiencia metabólica. 7. Uso de los polimorfismos según la reivindicación 6, donde la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) 8. Uso de un kit que comprende las sondas y cebadores necesarios para la determinación de la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, para determinar la eficiencia metabólica.
Los productos y servicios protegidos por este registro son:
C12Q 1/68
Descripciones:
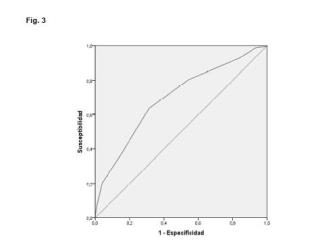
+ ES-2958596_A1 MARCADORES DE EFICIENCIA METABÓLICA CAMPO DE LA INVENCIÓN La presente invención se enmarca en el campo general de la genética deportiva, y en particular se refiere a un método para determinar la eficiencia metabólica en un individuo. ESTADO DE LA TÉCNICA El rendimiento de resistencia en un deportista está relacionado con un fenotipo complejo, influenciado por una serie de factores intrínsecos y extrínsecos. Entre los factores intrínsecos, la probabilidad de convertirse en un atleta de resistencia está influenciado por la composición de fibras musculares esqueléticas del atleta, gasto cardíaco máximo y consumo de oxígeno (VO2máx.) durante el ejercicio, la eficiencia metabólica y masa total de hemoglobina. Curiosamente, la mayoría de estos rasgos están fuertemente influenciados por la genética, mientras que algunos de ellos pueden modificarse positivamente con ejercicios de resistencia capacitación. Se puede considerar por tanto que, el estatus de un atleta de resistencia viene determinado por la combinación óptima de predisposición genética y acondicionamiento físico adecuado (Eynon, et al. Genes for elite power and sprint performance: ACTN3 leads the way. Sports Med. 43 (9) : 803-817. doi:10.1007/s40279-013-0059-4. PMID:23681449) . En cuanto a la predisposición genética de un individuo para ser deportista de resistencia, hay estudios que demuestran que al menos 120 marcadores genéticos están vinculados a esta condición y casi todos los cromosomas contienen al menos 1 gen asociado con el deporte rendimiento. A partir de estos marcadores genéticos de rendimiento, más de 70 se asocian con actividades deportivas de resistencia. (Ahmetov, I.I.et al. 2015. Chapter Six - Current progress in sports genomics. Adv. Clin. Chem. 70: 247-314. doi:10.1016/bs.acc.2015.03.003. PMID:26231489) . Existen diferentes estudios sobre las variantes genéticas que influyen en el rendimiento, alguno de ellos, demostraron que varios genes asociados con la eficiencia metabólica podrían implicar una mejora en la capacidad de resistencia a través de la disminución del estrés oxidativo. En este sentido, la solicitud de patente WO2018036495 y la solicitud de atente WO2011094815 describen el uso de diferentes polimorfismos o Single Nucleotide Polymorphisms (SNPs) para determinar diferentes cualidades de un sujeto para el deporte. Entre los polimorfismos estudiados en estas solicitudes de patente, relacionan los polimorfismos del gen de la adenosina monofosfato desaminasa 1 (AMPD1) c.34C>T (rs17602729) , el receptor gamma 1-alfa activado por el proliferador de peroxisomas (PGC1a) c.1444G>A (rs8192678) , y en gen regulador de la homeostasis del hierro (HFE) c.187C>G (rs1799945) y c.845G>A (rs1800562) con la eficiencia metabólica. Sin embargo, muchos de los polimorfismos estudiados no son determinantes. Existe pues la necesidad de proporcionar un método genético para determinar la eficiencia metabólica cuyo panel de marcadores sea altamente concluyente, de tal forma que, además de determinar la eficiencia metabólica en un individuo, sirva para pronosticar el talento deportivo y crear entrenamientos personalizados aumentando de esta manera, el potencial del deportista. DESCRIPCIÓN BREVE DE LA INVENCIÓN La presente invención soluciona el problema descrito en el estado de la técnica ya que proporciona un método para la determinación de la eficiencia metabólica en un individuo basado en el estudio de variantes genéticas en varios perfiles poligénicos. Así pues, en un primer aspecto, la presente invención se refiere a un método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) o, - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) es indicativo de eficiencia metabólica. En la presente por "eficiencia metabólica" se refiere a la capacidad de resistencia del músculo, a la capacidad del metabolismo hepático y a la recuperación del músculo ante los esfuerzos. En la presente invención por "capacidad de resistencia del músculo" se refiere a la capacidad que tiene un músculo o grupo de músculos para sostener contracciones repetidas contra una resistencia durante un período de tiempo prolongado. Por lo tanto, cuando la presente invención se refiere a que el músculo tiene capacidad de resistencia, se refiere a que es capaz de soportar repetidas contracciones durante un periodo de iempo. Donde el periodo de tiempo prolongado es de al menos 1 minuto, preferiblemente de al menos 10 minutos, preferiblemente de al menos 15 minutos. En la presente invención por "capacidad de metabolismo hepático" ser refiere a la capacidad que tiene el hígado de detoxificar los radicales libres y peróxidos de hidrógeno que se producen tras un esfuerzo muscular, dando lugar a una rápida recuperación sistémica u homeostasis. En la presente invención cuando se refiere a capacidad de metabolismo hepático, se refiere a que el hígado detoxifica y elimina con eficacia y eficiencia los radicales libres y peróxidos de hidrógeno producidos tras un esfuerzo muscular. En la presente invención por recuperación del músculo ante los esfuerzos se refiere a una óptima efectividad metabólica, entendida como resistencia muscular y una óptima capacidad de detoxificación sistémica, entendida como eliminación sistémica de los productos de desechos ante esfuerzos musculares de larga duración. En la presente invención, eficiencia metabólica es indicativa de talento deportivo. En la presente invención por "talento deportivo" se refiere al conjunto de aptitudes o características genéticamente dependientes que posee una persona para poder alcanzar altos resultados deportivos. En una realización particular, el talento deportivo está asociado a deportes de resistencia. En la presente invención por "resistencia" se refiere a aquellos deportes que requieren el mantenimiento de un esfuerzo de manera eficaz durante el mayor tiempo posible, este tipo de deportes requiere de una alta capacidad cardiaca, pulmonar y circulatoria. En una realización particular, el deporte de resistencia se refiere a resistencia aeróbica. En otra realización particular, el deporte de resistencia se refiere a resistencia muscular. Ejemplos de deportes de resistencia son el ciclismo, atletas de fondo (distancias superiores a 3000 m) , atletas de trekking, triatletas, tenistas, entre otros. En la presente invención por "polimorfismos óptimos" se refiere a los descritos en la tabla 1 y 2 con una puntuación genotípica igual a 2. En la presente invención, los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 .506-1G>A (rs3892097) y GSTP1 c.313A>G (rs1695) son polimorfismos de nucleótido único (SNP) . En la presente invención por "presencia conjunta" se refiere a que todas las variantes polimórficas en la combinación de polimorfismos presentan el polimorfismo óptimo. En otra realización particular, la muestra aislada obtenida de un individuo es una muestra de sangre, mucosa bucal o saliva. En otra realización particular la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. En otra realización particular, los genes se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , En otra realización particular, la genotipificación mediante PCR-SNPE, fue seguida de un ensayo de extensión de base única de sonda-cebadores. En otro aspecto, la presente invención se refiere a un método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1-1+, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y PGC1 c.1444 G>A (rs8192678) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y GSTM1 +/- o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- o - PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) y HFE c.845G>A (rs1800562) o - CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) y HFE c.845G>A (rs1800562) es indicativo de eficiencia metabólica. En otro aspecto, la presente invención se refiere al uso de los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- como marcadores de eficiencia metabólica. En una realización particular, la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) En otra realización particular, la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y PGC1 c.1444 G>A (rs8192678) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y GSTM1 +/- o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- o - PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) y HFE c.845G>A (rs1800562) o - CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) y HFE c.845G>A (rs1800562) En la presente invención, eficiencia metabólica es indicativa de talento deportivo. En una realización particular, el talento deportivo está asociado a deportes de resistencia. En otra realización particular, la muestra aislada obtenida de un individuo es una muestra de sangre, mucosa bucal o saliva. En otra realización particular la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. En otra realización preferente, los genes se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , En otra realización preferente, la genotipificación mediante PCR-SNPE, fue seguida de un ensayo de extensión de base única de sondacebadores. En otro aspecto, la presente invención se refiere al uso de un kit que comprende las sondas y cebadores necesarios para la determinación de la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, para determinar la eficiencia metabólica. DESCRIPCIÓN DE LOS DIBUJOS Fig. 1: Muestra los valores medios de TGS de los genes de eficiencia metabólica en: a) deportistas profesionales y sujetos no deportistas: *p<0, 001 y, b) deportistas de resistencia y jugadores de fútbol con respecto a sujetos no deportistas; |p<0.001; Hp<0.001. Fig.2: Muestra la distribución TGS de los genes de eficiencia metabólica en: a) deportistas profesionales y sujetos no deportistas y b) deportistas de resistencia y jugadores de fútbol con respecto a sujetos no deportistas. Fig. 3: Muestra la Curva ROC que resume la capacidad de TGS de los genes de la eficiencia metabólica para distinguir a los deportistas profesionales de la población no deportista, es decir, discrimina la probabilidad de ser deportista de élite frente a población no deportista. Fig. 4: Muestra la Curva ROC que resume la capacidad de TGS de los genes de la eficiencia metabólica para distinguir a los deportistas de resistencia de los futbolistas profesionales, es decir, discrimina la probabilidad de ser deportista de resistencia frente a futbolista profesional. DESCRIPCIÓN DETALLADA DE LA INVENCIÓN Se preparó un estudio en el que se determinaron las frecuencias genotípicas y alélicas en 414 sujetos: - 292 atletas profesionales: o 160 deportistas de resistencia o 132 futbolistas profesionales - 122 sujetos no deportistas. Los no deportistas fueron emparejados por edad con los deportistas de élite, siendo el criterio de inclusión que no fueran fumadores, y que no padecieran enfermedades crónicas o agudas, ni obesidad, en el momento de la toma de muestras. La participación y aceptación de inclusión de los participantes se obtuvo mediante la firma del documento de consentimiento informado. El protocolo de estudio fue aprobado por el Comité Ético de la Universidad de Valladolid y se ajustaba a la Declaración de Helsinki para la Investigación en Humanos de 1964 (última modificación en 2013) . Con el fin de investigar el papel de diferentes variantes genéticas relacionadas con el estado de los deportistas de élite, se seleccionaron los siguientes perfiles poligénicos con polimorfismos funcionales en los siguientes genes diana: - AMPD1 c.34C>T (rs17602729) - PGC1ac.1444 G>A (rs8192678) - HFE c.187C>G (rs1799945) - HFE c.845G>A (rs1800562) - CYP2D6 c.506-1G>A (rs3892097) - GSTM1 +/-- GSTP1 c.313A>G (rs1695) - GSTT +/- La extracción de ADN se realizó mediante frotis oral con hisopos SARSTED, y se mantuvo en frío hasta su extracción en el laboratorio. A continuación, el ADN genómico de las muestras de mucosa oral se extrajo mediante extracción automática utilizando el equipo QIACube (QIAGEN, Venlo, Holanda) , con un rendimiento de concentración de ADN de 25-40 ng/ml, mantenidas en solución en un volumen de 100 microlitros a -20°C hasta el genotipado. Todos los genes seleccionados se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , (Vallone PM, Just RS, Coble MD, Butler JM, Parsons TJ. A multiplex allele-specific primer extension assay for forensically informative SNPs distributed throughout the mitochondrial genome. Int J Legal Med. 2004;118 (3) :147-57. https://pubmed.ncbi.nlm.nih.gov/14760491) una PCR multiplex para la amplificación de secuencias específicas, seguido de un ensayo de extensión de base única de sondacebadores utilizando el kit comercial SNaPshot. (Applied Biosystems, Foster City, CA) en el instrumento de PCR en tiempo real QuantStudio5 (QS5) (Thermofisher, CA) . La influencia combinada de cada uno de los polimorfismos en la eficiencia metabólica se calculó siguiendo el procedimiento de Williams y Folland. Los polimorfismos candidatos son bialélicos a excepción del polimorfismo del gen GSTM en el que el genotipo óptimo presenta un valor de 1 y, de acuerdo con investigaciones previas sobre la eficiencia metabólica, los polimorfismos se calificaron dando la siguiente puntuación: - 2 puntos a un genotipo (GS) óptimo (homocigotos favorables) - 1 punto a los heterocigotos y - 0 puntos a homocigotos no favorables. Las puntuaciones obtenidas en cada polimorfismo genético se sumaron para obtener una puntuación de genotipo total perfecta que representa el genotipo óptimo, que fue para el perfil de eficiencia metabólica de 15 a.u. (rango 0-15 au) . Finalmente, este valor se transformó en una escala de 0-100 a.u. para facilitar la interpretación mediante la puntuación total del genotipo (TGS) , como sigue. En base a dicha fórmula, un TGS de 100 u.a. representa el "perfil perfecto", mientras que un TGS de 0 u.a. sería el "peor perfil" posible ya que todos los GS tienen una puntuación de 0 u.a. Finalmente se evaluó la distribución del TGS entre los diferentes perfiles de deportistas profesionales entre los que se encuentran deportistas de élite de resistencia, futbolistas profesionales y población no deportista. El uso de herramientas estadísticas para medir los resultados encontrados se realizó con promedio estadístico y la escala curtosis. Se probó que todos los polimorfismos usados cumplían el equilibrio de Hardy-Weinberg (HWE) usando pruebas de x2. La probabilidad de tener un genotipo "óptimo" para este perfil genético entre deportistas profesionales y población no deportista se calculó mediante la prueba de x2 con un a fijo de 0.05. Las frecuencias genotípicas de los polimorfismos en el perfil de eficiencia metabólica se compararon entre los valores de referencia de los deportistas profesionales y no deportistas, utilizando una prueba de x2 con a fijo de 0, 05 para comprobar la similitud del perfil en el individuo analizado. La capacidad de TGS para distinguir correctamente a los deportistas profesionales potenciales de los no deportistas (0 = atleta profesional, 1 = no tleta) se evaluó mediante curvas de características operativas del receptor (ROC) para establecer un valor de corte en este perfil. Para ello se calculó el área bajo la curva ROC (AUC) con intervalos de confianza del 95% (IC 95%) . Finalmente, se utilizó un modelo de regresión logística binaria para estudiar la relación entre TGS y el estatus de deportista profesional en comparación con los no deportistas y establecer a su vez un valor de eficiencia metabólica y recuperación sistémica de esfuerzos. El análisis en conjunto de los ocho polimorfismos del perfil de eficiencia metabólica (AMPD1 c.34C>T (rs17602729) , PGC1a c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, STP1 c.313A>G (rs1695) y GSTT +/-) presentó una puntuación de TGS total: - TGS en deportistas profesionales: 59.12 u.a. (±10.53 u.a.) , curtosis estadística: -0.228 (±0.252) - TGS en deportistas de resistencia: 59.83 u.a (±10.01 u.a.) , curtosis estadística: -0.249 (±0.402) - TGS en futbolistas profesionales: 58.27 u.a (±10.91 u.a.) , curtosis estadística: -0.230 (±0.315) - TGS población no deportista: 52.61 u.a. (±10.46 u.a.) curtosis estadística: 0.147 (±0.383) . Estos TGS presentaron diferencias estadísticas entre los deportistas de resistencia y población no deportista (p<0.001) (Figura 1a) . Entre las dos modalidades de deportistas, no se mostraron diferencias en el puntaje total de los polimorfismos de eficiencia metabólica, (p=0.684) , pero sí entre ambas modalidades deportivas y la población no deportista (p<0.001) (Figura 1b) . Los datos mostraron que las diferencias en las distribuciones de los valores de TGS entre no deportistas y deportistas profesionales fueron estadísticamente significativas (p<0.001) (Figura 2a) , al igual que entre ambos grupos con no deportistas (p<0.001) (Figura 2b) . El análisis ROC mostró una precisión discriminatoria significativa del TGS en la identificación de deportistas profesionales (AUC = 0.674; 95% CI: 0.617-0.731; p < 0.001) (sensibilidad = 0.812, especificidad = 0.557) (Fig. 3) . El valor TGS correspondiente n este punto fue de 49.99 u.a. El análisis de regresión logística binaria mostró que los sujetos con un TGS más alto de 49.99 u.a. tenían un OR de 3.310 (95% CI: 2.084-5.528; p < 0.001) de ser atletas profesionales, en comparación con aquellos con un TGS por debajo de este valor. Los deportistas de resistencia de élite mostraron una OR en el punto de corte en comparación con la población no deportista de 4.148 (95% CI: 2.376 7.240; p < 0.001) y los jugadores de fútbol profesional, en comparación con los sujetos no deportistas, tenían una OR de 2.612 (95% CI: 1.519-4.491; p = 0, 001) . La distribución del genotipo de polimorfismos de eficiencia metabólica en el grupo de atletas profesionales en comparación con la población de no atletas fue estadísticamente significativa para el polimorfismo HFE c.187C>G (p = 0.001) , mostrando una mayor frecuencia en el genotipo "óptimo" en atletas (GG 5.80%) que la población no deportista (GG 0.00%) , así como en el genotipo AMPD1 CC (94.20% vs. 62, 10% respectivamente; p = 0.006) y el polimorfismo CYP2D6 c.506-1G>A "óptimo" (p < 0.001) (Tabla 1) . Por otro lado, los datos demostraron que, en el polimorfismo HFE c.187C>G existían diferencias estadísticamente significativas entre ambos grupos de deportistas de resistencia (p < 0.001) y futbolistas profesionales (p = 0.013) con respecto a la población no deportista. En cuanto al polimorfismo AMPD1, también se encontraron diferencias entre deportistas de resistencia, y la población no deportista (p = 0.010) y futbolistas profesionales y no deportistas (p = 0.014) . Igualmente, en el polimorfismo CYP2D6 c.506-1G>A se encontraron diferencias estadísticamente significativas entre ambos grupos de deportistas de resistencia y futbolistas profesionales y la población no deportista (p < 0.001) (tabla 2) . Entre los dos grupos de deportistas profesionales (jugadores de fútbol y de resistencia) , se encontraron resultados estadísticamente significativos en los polimorfismos de HFE c.187C>G mostrando genotipos más favorables en los atletas de resistencia frente a los jugadores de fútbol (p = 0.001) , así como en el polimorfismo GSTP c.313A>G (p = 0.014) , sin embargo en el polimorfismo CYP2D6 c.506-1G>A se presentaron genotipos más favorables en los futbolistas profesionales frente a los atletas de resistencia (p = 0.002) (Tabla 2) . Tabla 1. Distribución de genotipos en atletas profesionales y no atletas de polimorfismos de eficiencia metabolizadora Tabla 2 Para comprobar los perfiles que presentaban un TGS predictivo de eficiencia metabólica entre la población deportista y no deportista, se analizaron combinaciones de 4 en 4 polimorfismos, y de tres en tres polimorfismos. Para ello, las puntuaciones obtenidas en cada polimorfismo genético se sumaron para obtener una puntuación de genotipo total perfecto que representa el genotipo óptimo, y que fue de 8 u.a (rango 0-8 u.a) . Finalmente, este valor se transformó en una escala de 0-100 u.a para facilitar la interpretación mediante la puntuación total del genotipo (TGS) en esta combinación de 4 polimorfismos. Se realizó el mismo cálculo para las combinaciones de tres polimorfismos. 1. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- Los resultados de la combinación de estos cuatro polimorfismos mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 64.31 u.a.) frente a la población no deportista (TGS = 55.85 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, igualmente hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 66.83 u.a.) frente a los futbolistas profesionales (TGS = 62.17 u.a.) (p=0.003) , Estos datos sugirieron que, la combinación de estos cuatro polimorfismos tuvo un valor predictivo positivo de eficiencia metabólica y pudiéndose discriminar dentro de las diferentes modalidades deportivas. 2. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/-y HFE c.187C>G (rs1799945) Los resultados muestran un valor de asociación positiva en los deportistas profesionales (TGS = 60.34 u.a.) frente a la población no deportista (TGS = 54.91 u.a.) (p=0.002) , al igual que dentro de los grupos de los deportistas, donde se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 58.14 u.a.) (p=0.013) , presentando discriminación en el TGS en estos polimorfismos entre las modalidades deportivas. 3. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron en estos polimorfismos un valor de asociación positiva en los deportistas profesionales (TGS = 74.64 u.a.) frente a la población no deportista (TGS = 69.56 u.a.) (p=0.005) , al contrario que lo observado en los grupos de los deportistas, en los que no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 76.33 u.a.) frente a los futbolistas profesionales (TGS = 73.20 u.a.) (p=0.124) , no presentando discriminación en el TGS en estos polimorfismos entre las modalidades deportivas. 4. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y HFE c.187C>G (rs1799945) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.34 u.a.) frente a la población no deportista (TGS = 54.91 u.a.) (p=0.002) . Dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 58.14 u.a.) (p=0.013) , lo que mostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 5. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFEc.187C>G (rs1799945) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 69.41 u.a.) frente a la población no deportista (TGS = 62.09 u.a.) (p<0.001) . Igualmente, dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 71.31 u.a.) frente a los futbolistas profesionales (TGS = 67.80 u.a.) (p=0.038) , lo que significa que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos, favoreciendo esta eficiencia metabólica en términos genéticos a los deportistas de resistencia. 6. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1ac.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 83.81 u.a.) frente a la población no deportista (TGS = 76.43 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con un TGS de 84.70 u.a. en los deportistas de resistencia frente a un TGS = 83.04 u.a en los futbolistas profesionales (p=0.335) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 7. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 71.15 u.a.) frente a la población no deportista (TGS = 62.91 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.88 u.a.) frente a los futbolistas (TGS = 68.84 u.a.) (p=0.008) , lo que significa que dentro de las diferentes modalidades deportivas existió discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 8. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y CYP2D6 c.506-1G>A (rs3892097) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 78.48 u.a.) frente a la población no deportista (TGS = 71.51 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 79.24 u.a.) frente a los futbolistas profesionales (TGS = 77.84 u.a.) (p=0.441) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 9. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-y GSTP1 c.313A>G (rs1695) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.02 u.a.) frente a la población no deportista (TGS = 46.31 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.58 u.a.) frente a los futbolistas profesionales (TGS = 49.14 u.a.) (p<0.001) , lo que significó que dentro de las diferentes modalidades deportivas existía discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 10. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 50.92 u.a.) frente a la población no deportista (TGS = 42.11 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 54.35 u.a.) frente a los futbolistas (TGS = 48.01 u.a.) (p=0.004) , lo que demostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 11. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.09 u.a.) frente a la población no deportista (TGS = 48.97 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.72 u.a.) frente a los futbolistas profesionales (TGS = 57.85 u.a.) (p=0.006) , lo que demostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 12. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 67.41 u.a.) frente a la población no deportista (TGS = 57.58 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 68.08 u.a.) frente a los futbolistas profesionales (TGS = 66.85 u.a.) (p=0.505) , lo que significó que dentro de las diferentes modalidades deportivas no hubo discriminación en el TGS en estos polimorfismos. 13. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.25 u.a.) frente a la población no deportista (TGS = 47.02 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 59.82 u.a.) frente a los futbolistas profesionales (TGS = 53.21 u.a.) (p=0.001) , lo que significó que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 14. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 72.74 u.a.) frente a la población no deportista (TGS = 62.50 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.54 u.a.) frente a los futbolistas profesionales (TGS = 72.06 u.a.) (p=0.351) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 15. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , GSTT+/-y PGC1 c.1444 G>A (rs8192678) . Los resultados muestran un valor de asociación positiva en los deportistas profesionales (TGS = 63.57 u.a.) frente a la población no deportista (TGS = 55.63 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 65.17 u.a.) frente a los futbolistas profesionales (TGS = 62.21 u.a.) (p=0.138) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 16. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) . Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.25 u.a.) frente a la población no deportista (TGS = 55.63 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 59.82 u.a.) frente a los futbolistas profesionales (TGS = 53.21 u.a.) (p=0.001) , lo que significó que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. En estas combinaciones de los 4 polimorfismos en la eficiencia metabólica, se mostró discriminación mediante TGS entre deportistas y población no deportista en todas las combinaciones posibles, presentando a su vez dentro de los dos grupos de deportistas el grupo de resistencia una mayor puntuación TFS en los marcadores seleccionados, presentando mejor selección adaptativa en términos genéticos en los polimorfismos implicados en eficiencia metabólica. Se estimó que la combinación más predictiva de deportistas y la discriminación entre el grupo de resistencia y futbolistas profesionales fue la combinación de cuatro polimorfismos, como se ha comprobado anteriormente, debido a que supera el puntaje del 50% del total, lo que otorga validez a esta estimación frente a las combinaciones de menos genes debido a la probabilidad de predecir falsos positivos en el perfil genético de eficiencia metabólica Para ello, se trabajó con diversas combinaciones para mostrar este valor predictivo en los TGS de únicamente 3 polimorfismos diferentes; 17. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , PGC1 c.1444 G>A (rs8192678) . Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 65.40 u.a.) frente a la población no deportista (TGS = 56.55 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 67.28 u.a.) frente a los futbolistas (TGS = 63.85 u.a.) (p=0.069) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 18. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.04 u.a.) frente a la población no deportista (TGS = 41.53 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo iferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 57.57 u.a.) (p=0.130) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 19. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , y GSTM1 +/- Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 59.64 u.a.) frente a la población no deportista (TGS = 51.31 u.a.) (p=0.003) . Igualmente, dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 68.39 u.a.) frente a los futbolistas profesionales (TGS = 52.22 u.a.) (p<0.001) , lo que significó que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia. 20. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , y GSTP1 c.313A>G (rs1695) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 71.59 u.a.) frente a la población no deportista (TGS = 68.29 u.a.) (p=0.448) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.60 u.a.) frente a los futbolistas profesionales (TGS = 69.89 u.a.) (p=0.398) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 21. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.71 u.a.) frente a la población no deportista (TGS = 43.60 u.a.) (p=0.003) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.76 u.a.) frente a los futbolistas profesionales (TGS = 50.27 u.a.) (p=0.058) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 22. Polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1a c.1444G>A (rs8192678) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.98 u.a.) frente a la población no deportista (TGS = 53.13 u.a.) (p=0.219) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.93 u.a.) frente a los futbolistas (TGS = 56.94 u.a.) (p=0.436) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 23. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 36.60 u.a.) frente a la población no deportista (TGS = 33.60 u.a.) (p=0.127) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 38.01 u.a.) frente a los futbolistas (TGS = 35.87 u.a.) (p=0.195) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 24. Polimorfismos PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 36.52 u.a.) frente a la población no deportista (TGS = 30.05 u.a.) (p<0.001) . Al igual que se observó dentro de los grupos de los deportistas, con diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 38.22 u.a.) frente a los futbolistas (TGS = 34.46 u.a.) (p=0.024) , lo que significó que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos. 25. Polimorfismos CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 62.95 u.a.) frente a la población no deportista (TGS = 55.73 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo iferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.39 u.a.) frente a los futbolistas (TGS = 63.63 u.a.) (p=0.653) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 26. Polimorfismos GTSM1 +/-, AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 45.34 u.a.) frente a la población no deportista (TGS = 42.13 u.a.) (p=0.098) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 44.62 u.a.) frente a los futbolistas (TGS = 46.21 u.a.) (p=0.350) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 27. Polimorfismos GSTP1 c.313A>G (rs1695) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.36 u.a.) frente a la población no deportista (TGS = 50.54 u.a.) (p=0.173) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 54.73 u.a.) frente a los futbolistas (TGS = 52.51 u.a.) (p=0.527) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 28. Polimorfismos GSTT+/-, AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 49.91 u.a.) frente a la población no deportista (TGS = 46.58 u.a.) (p=0.69) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 49.57 u.a.) frente a los futbolistas (TGS = 50.50 u.a.) (p=0.632) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 29. Polimorfismos PGC1a c.1444G>A (rs8192678) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 55.98 u.a.) frente a la población no deportista (TGS = 53.16 u.a.) (p=0.310) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 56.43 u.a.) frente a los futbolistas (TGS = 55.44 u.a.) (p=0.496) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. Como se puede apreciar, la combinación de 3 polimorfismos no recoge toda la discriminación en el TGS que sí se observa en todas las combinaciones de 4 polimorfismos entre los deportistas y la población no deportista. Esta combinación de 3 polimorfismos no presenta la capacidad discriminatoria entre grupos debido a la falta de potencia establecida, con una probabilidad alta de predecir falsos positivos en el perfil genético de eficiencia metabólica.
Publicaciones:
ES2958596 (12/02/2024) - A1 Solicitud de patente con informe sobre el estado de la técnica
Eventos:
En fecha 15/07/2022 se realizó Registro Instancia de Solicitud
En fecha 15/07/2022 se realizó Admisión a Trámite
En fecha 15/07/2022 se realizó 1001P_Comunicación Admisión a Trámite
En fecha 28/07/2022 se realizó Superado examen de oficio
En fecha 12/04/2023 se realizó Realizado IET
En fecha 14/04/2023 se realizó 1109P_Comunicación Traslado del IET
En fecha 12/02/2024 se realizó Publicación Solicitud
En fecha 12/02/2024 se realizó Publicación Folleto Solicitud con IET (A1)
En fecha 09/04/2024 se realizó PETEX_Petición de examen sustantivo
Pagos:
15/07/2022 - Pago Tasas IET
+ ES-2958596_A11.- Método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1-1+, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) o, - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) es indicativo de eficiencia metabólica. 2. Método según la reivindicación 1, donde la eficiencia metabólica es indicativa de talento deportivo. 3. Método según la reivindicación 2, donde el talento deportivo está asociado a deportes de resistencia. 4. Método según cualquiera de las reivindicaciones anteriores, donde la muestra es una muestra de sangre, mucosa bucal o saliva. 5. Método según cualquiera de las reivindicaciones anteriores donde la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. 6. Uso de los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- como marcadores de eficiencia metabólica. 7. Uso de los polimorfismos según la reivindicación 6, donde la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) 8. Uso de un kit que comprende las sondas y cebadores necesarios para la determinación de la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, para determinar la eficiencia metabólica.
Los productos y servicios protegidos por este registro son:
C12Q 1/68
Descripciones:
+ ES-2958596_A1 MARCADORES DE EFICIENCIA METABÓLICA CAMPO DE LA INVENCIÓN La presente invención se enmarca en el campo general de la genética deportiva, y en particular se refiere a un método para determinar la eficiencia metabólica en un individuo. ESTADO DE LA TÉCNICA El rendimiento de resistencia en un deportista está relacionado con un fenotipo complejo, influenciado por una serie de factores intrínsecos y extrínsecos. Entre los factores intrínsecos, la probabilidad de convertirse en un atleta de resistencia está influenciado por la composición de fibras musculares esqueléticas del atleta, gasto cardíaco máximo y consumo de oxígeno (VO2máx.) durante el ejercicio, la eficiencia metabólica y masa total de hemoglobina. Curiosamente, la mayoría de estos rasgos están fuertemente influenciados por la genética, mientras que algunos de ellos pueden modificarse positivamente con ejercicios de resistencia capacitación. Se puede considerar por tanto que, el estatus de un atleta de resistencia viene determinado por la combinación óptima de predisposición genética y acondicionamiento físico adecuado (Eynon, et al. Genes for elite power and sprint performance: ACTN3 leads the way. Sports Med. 43 (9) : 803-817. doi:10.1007/s40279-013-0059-4. PMID:23681449) . En cuanto a la predisposición genética de un individuo para ser deportista de resistencia, hay estudios que demuestran que al menos 120 marcadores genéticos están vinculados a esta condición y casi todos los cromosomas contienen al menos 1 gen asociado con el deporte rendimiento. A partir de estos marcadores genéticos de rendimiento, más de 70 se asocian con actividades deportivas de resistencia. (Ahmetov, I.I.et al. 2015. Chapter Six - Current progress in sports genomics. Adv. Clin. Chem. 70: 247-314. doi:10.1016/bs.acc.2015.03.003. PMID:26231489) . Existen diferentes estudios sobre las variantes genéticas que influyen en el rendimiento, alguno de ellos, demostraron que varios genes asociados con la eficiencia metabólica podrían implicar una mejora en la capacidad de resistencia a través de la disminución del estrés oxidativo. En este sentido, la solicitud de patente WO2018036495 y la solicitud de atente WO2011094815 describen el uso de diferentes polimorfismos o Single Nucleotide Polymorphisms (SNPs) para determinar diferentes cualidades de un sujeto para el deporte. Entre los polimorfismos estudiados en estas solicitudes de patente, relacionan los polimorfismos del gen de la adenosina monofosfato desaminasa 1 (AMPD1) c.34C>T (rs17602729) , el receptor gamma 1-alfa activado por el proliferador de peroxisomas (PGC1a) c.1444G>A (rs8192678) , y en gen regulador de la homeostasis del hierro (HFE) c.187C>G (rs1799945) y c.845G>A (rs1800562) con la eficiencia metabólica. Sin embargo, muchos de los polimorfismos estudiados no son determinantes. Existe pues la necesidad de proporcionar un método genético para determinar la eficiencia metabólica cuyo panel de marcadores sea altamente concluyente, de tal forma que, además de determinar la eficiencia metabólica en un individuo, sirva para pronosticar el talento deportivo y crear entrenamientos personalizados aumentando de esta manera, el potencial del deportista. DESCRIPCIÓN BREVE DE LA INVENCIÓN La presente invención soluciona el problema descrito en el estado de la técnica ya que proporciona un método para la determinación de la eficiencia metabólica en un individuo basado en el estudio de variantes genéticas en varios perfiles poligénicos. Así pues, en un primer aspecto, la presente invención se refiere a un método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) o, - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) es indicativo de eficiencia metabólica. En la presente por "eficiencia metabólica" se refiere a la capacidad de resistencia del músculo, a la capacidad del metabolismo hepático y a la recuperación del músculo ante los esfuerzos. En la presente invención por "capacidad de resistencia del músculo" se refiere a la capacidad que tiene un músculo o grupo de músculos para sostener contracciones repetidas contra una resistencia durante un período de tiempo prolongado. Por lo tanto, cuando la presente invención se refiere a que el músculo tiene capacidad de resistencia, se refiere a que es capaz de soportar repetidas contracciones durante un periodo de iempo. Donde el periodo de tiempo prolongado es de al menos 1 minuto, preferiblemente de al menos 10 minutos, preferiblemente de al menos 15 minutos. En la presente invención por "capacidad de metabolismo hepático" ser refiere a la capacidad que tiene el hígado de detoxificar los radicales libres y peróxidos de hidrógeno que se producen tras un esfuerzo muscular, dando lugar a una rápida recuperación sistémica u homeostasis. En la presente invención cuando se refiere a capacidad de metabolismo hepático, se refiere a que el hígado detoxifica y elimina con eficacia y eficiencia los radicales libres y peróxidos de hidrógeno producidos tras un esfuerzo muscular. En la presente invención por recuperación del músculo ante los esfuerzos se refiere a una óptima efectividad metabólica, entendida como resistencia muscular y una óptima capacidad de detoxificación sistémica, entendida como eliminación sistémica de los productos de desechos ante esfuerzos musculares de larga duración. En la presente invención, eficiencia metabólica es indicativa de talento deportivo. En la presente invención por "talento deportivo" se refiere al conjunto de aptitudes o características genéticamente dependientes que posee una persona para poder alcanzar altos resultados deportivos. En una realización particular, el talento deportivo está asociado a deportes de resistencia. En la presente invención por "resistencia" se refiere a aquellos deportes que requieren el mantenimiento de un esfuerzo de manera eficaz durante el mayor tiempo posible, este tipo de deportes requiere de una alta capacidad cardiaca, pulmonar y circulatoria. En una realización particular, el deporte de resistencia se refiere a resistencia aeróbica. En otra realización particular, el deporte de resistencia se refiere a resistencia muscular. Ejemplos de deportes de resistencia son el ciclismo, atletas de fondo (distancias superiores a 3000 m) , atletas de trekking, triatletas, tenistas, entre otros. En la presente invención por "polimorfismos óptimos" se refiere a los descritos en la tabla 1 y 2 con una puntuación genotípica igual a 2. En la presente invención, los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 .506-1G>A (rs3892097) y GSTP1 c.313A>G (rs1695) son polimorfismos de nucleótido único (SNP) . En la presente invención por "presencia conjunta" se refiere a que todas las variantes polimórficas en la combinación de polimorfismos presentan el polimorfismo óptimo. En otra realización particular, la muestra aislada obtenida de un individuo es una muestra de sangre, mucosa bucal o saliva. En otra realización particular la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. En otra realización particular, los genes se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , En otra realización particular, la genotipificación mediante PCR-SNPE, fue seguida de un ensayo de extensión de base única de sonda-cebadores. En otro aspecto, la presente invención se refiere a un método in vitro para determinar la eficiencia metabólica en un individuo que comprende identificar la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1-1+, GSTP1 c.313A>G (rs1695) y GSTT +/- en una muestra aislada obtenida de un individuo, donde la presencia conjunta de los polimorfismos óptimos en combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y PGC1 c.1444 G>A (rs8192678) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y GSTM1 +/- o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- o - PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) y HFE c.845G>A (rs1800562) o - CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) y HFE c.845G>A (rs1800562) es indicativo de eficiencia metabólica. En otro aspecto, la presente invención se refiere al uso de los polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- como marcadores de eficiencia metabólica. En una realización particular, la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos: - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFE c.187C>G (rs1799945) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1a c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT +/- y CYP2D6 c.506-1G>A (rs3892097) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTP1 c.313A>G (rs1695) , o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/-, o - AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) , PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTT +/-, PGC1 c.1444 G>A (rs8192678) , o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-, PGC1 c.1444 G>A (rs8192678) En otra realización particular, la eficiencia metabólica se determina mediante la presencia conjunta de los polimorfismos óptimos en la combinación de polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y PGC1 c.1444 G>A (rs8192678) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y GSTM1 +/- o - AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- o - PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) y HFE c.845G>A (rs1800562) o - CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) y HFE c.845G>A (rs1800562) En la presente invención, eficiencia metabólica es indicativa de talento deportivo. En una realización particular, el talento deportivo está asociado a deportes de resistencia. En otra realización particular, la muestra aislada obtenida de un individuo es una muestra de sangre, mucosa bucal o saliva. En otra realización particular la presencia o ausencia del polimorfismo óptimo es determinada mediante PCR. En otra realización preferente, los genes se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , En otra realización preferente, la genotipificación mediante PCR-SNPE, fue seguida de un ensayo de extensión de base única de sondacebadores. En otro aspecto, la presente invención se refiere al uso de un kit que comprende las sondas y cebadores necesarios para la determinación de la ausencia o presencia de los polimorfismos óptimos AMPD1 c.34C>T (rs17602729) , PGC1 c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/-, para determinar la eficiencia metabólica. DESCRIPCIÓN DE LOS DIBUJOS Fig. 1: Muestra los valores medios de TGS de los genes de eficiencia metabólica en: a) deportistas profesionales y sujetos no deportistas: *p<0, 001 y, b) deportistas de resistencia y jugadores de fútbol con respecto a sujetos no deportistas; |p<0.001; Hp<0.001. Fig.2: Muestra la distribución TGS de los genes de eficiencia metabólica en: a) deportistas profesionales y sujetos no deportistas y b) deportistas de resistencia y jugadores de fútbol con respecto a sujetos no deportistas. Fig. 3: Muestra la Curva ROC que resume la capacidad de TGS de los genes de la eficiencia metabólica para distinguir a los deportistas profesionales de la población no deportista, es decir, discrimina la probabilidad de ser deportista de élite frente a población no deportista. Fig. 4: Muestra la Curva ROC que resume la capacidad de TGS de los genes de la eficiencia metabólica para distinguir a los deportistas de resistencia de los futbolistas profesionales, es decir, discrimina la probabilidad de ser deportista de resistencia frente a futbolista profesional. DESCRIPCIÓN DETALLADA DE LA INVENCIÓN Se preparó un estudio en el que se determinaron las frecuencias genotípicas y alélicas en 414 sujetos: - 292 atletas profesionales: o 160 deportistas de resistencia o 132 futbolistas profesionales - 122 sujetos no deportistas. Los no deportistas fueron emparejados por edad con los deportistas de élite, siendo el criterio de inclusión que no fueran fumadores, y que no padecieran enfermedades crónicas o agudas, ni obesidad, en el momento de la toma de muestras. La participación y aceptación de inclusión de los participantes se obtuvo mediante la firma del documento de consentimiento informado. El protocolo de estudio fue aprobado por el Comité Ético de la Universidad de Valladolid y se ajustaba a la Declaración de Helsinki para la Investigación en Humanos de 1964 (última modificación en 2013) . Con el fin de investigar el papel de diferentes variantes genéticas relacionadas con el estado de los deportistas de élite, se seleccionaron los siguientes perfiles poligénicos con polimorfismos funcionales en los siguientes genes diana: - AMPD1 c.34C>T (rs17602729) - PGC1ac.1444 G>A (rs8192678) - HFE c.187C>G (rs1799945) - HFE c.845G>A (rs1800562) - CYP2D6 c.506-1G>A (rs3892097) - GSTM1 +/-- GSTP1 c.313A>G (rs1695) - GSTT +/- La extracción de ADN se realizó mediante frotis oral con hisopos SARSTED, y se mantuvo en frío hasta su extracción en el laboratorio. A continuación, el ADN genómico de las muestras de mucosa oral se extrajo mediante extracción automática utilizando el equipo QIACube (QIAGEN, Venlo, Holanda) , con un rendimiento de concentración de ADN de 25-40 ng/ml, mantenidas en solución en un volumen de 100 microlitros a -20°C hasta el genotipado. Todos los genes seleccionados se genotipificaron mediante análisis multiplex mediante Polymerase Chain Reaction-Single Nucleotide Primer Extension (PCR-SNPE) , (Vallone PM, Just RS, Coble MD, Butler JM, Parsons TJ. A multiplex allele-specific primer extension assay for forensically informative SNPs distributed throughout the mitochondrial genome. Int J Legal Med. 2004;118 (3) :147-57. https://pubmed.ncbi.nlm.nih.gov/14760491) una PCR multiplex para la amplificación de secuencias específicas, seguido de un ensayo de extensión de base única de sondacebadores utilizando el kit comercial SNaPshot. (Applied Biosystems, Foster City, CA) en el instrumento de PCR en tiempo real QuantStudio5 (QS5) (Thermofisher, CA) . La influencia combinada de cada uno de los polimorfismos en la eficiencia metabólica se calculó siguiendo el procedimiento de Williams y Folland. Los polimorfismos candidatos son bialélicos a excepción del polimorfismo del gen GSTM en el que el genotipo óptimo presenta un valor de 1 y, de acuerdo con investigaciones previas sobre la eficiencia metabólica, los polimorfismos se calificaron dando la siguiente puntuación: - 2 puntos a un genotipo (GS) óptimo (homocigotos favorables) - 1 punto a los heterocigotos y - 0 puntos a homocigotos no favorables. Las puntuaciones obtenidas en cada polimorfismo genético se sumaron para obtener una puntuación de genotipo total perfecta que representa el genotipo óptimo, que fue para el perfil de eficiencia metabólica de 15 a.u. (rango 0-15 au) . Finalmente, este valor se transformó en una escala de 0-100 a.u. para facilitar la interpretación mediante la puntuación total del genotipo (TGS) , como sigue. En base a dicha fórmula, un TGS de 100 u.a. representa el "perfil perfecto", mientras que un TGS de 0 u.a. sería el "peor perfil" posible ya que todos los GS tienen una puntuación de 0 u.a. Finalmente se evaluó la distribución del TGS entre los diferentes perfiles de deportistas profesionales entre los que se encuentran deportistas de élite de resistencia, futbolistas profesionales y población no deportista. El uso de herramientas estadísticas para medir los resultados encontrados se realizó con promedio estadístico y la escala curtosis. Se probó que todos los polimorfismos usados cumplían el equilibrio de Hardy-Weinberg (HWE) usando pruebas de x2. La probabilidad de tener un genotipo "óptimo" para este perfil genético entre deportistas profesionales y población no deportista se calculó mediante la prueba de x2 con un a fijo de 0.05. Las frecuencias genotípicas de los polimorfismos en el perfil de eficiencia metabólica se compararon entre los valores de referencia de los deportistas profesionales y no deportistas, utilizando una prueba de x2 con a fijo de 0, 05 para comprobar la similitud del perfil en el individuo analizado. La capacidad de TGS para distinguir correctamente a los deportistas profesionales potenciales de los no deportistas (0 = atleta profesional, 1 = no tleta) se evaluó mediante curvas de características operativas del receptor (ROC) para establecer un valor de corte en este perfil. Para ello se calculó el área bajo la curva ROC (AUC) con intervalos de confianza del 95% (IC 95%) . Finalmente, se utilizó un modelo de regresión logística binaria para estudiar la relación entre TGS y el estatus de deportista profesional en comparación con los no deportistas y establecer a su vez un valor de eficiencia metabólica y recuperación sistémica de esfuerzos. El análisis en conjunto de los ocho polimorfismos del perfil de eficiencia metabólica (AMPD1 c.34C>T (rs17602729) , PGC1a c.1444 G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) , CYP2D6 c.506-1G>A (rs3892097) , GSTM1 +/-, STP1 c.313A>G (rs1695) y GSTT +/-) presentó una puntuación de TGS total: - TGS en deportistas profesionales: 59.12 u.a. (±10.53 u.a.) , curtosis estadística: -0.228 (±0.252) - TGS en deportistas de resistencia: 59.83 u.a (±10.01 u.a.) , curtosis estadística: -0.249 (±0.402) - TGS en futbolistas profesionales: 58.27 u.a (±10.91 u.a.) , curtosis estadística: -0.230 (±0.315) - TGS población no deportista: 52.61 u.a. (±10.46 u.a.) curtosis estadística: 0.147 (±0.383) . Estos TGS presentaron diferencias estadísticas entre los deportistas de resistencia y población no deportista (p<0.001) (Figura 1a) . Entre las dos modalidades de deportistas, no se mostraron diferencias en el puntaje total de los polimorfismos de eficiencia metabólica, (p=0.684) , pero sí entre ambas modalidades deportivas y la población no deportista (p<0.001) (Figura 1b) . Los datos mostraron que las diferencias en las distribuciones de los valores de TGS entre no deportistas y deportistas profesionales fueron estadísticamente significativas (p<0.001) (Figura 2a) , al igual que entre ambos grupos con no deportistas (p<0.001) (Figura 2b) . El análisis ROC mostró una precisión discriminatoria significativa del TGS en la identificación de deportistas profesionales (AUC = 0.674; 95% CI: 0.617-0.731; p < 0.001) (sensibilidad = 0.812, especificidad = 0.557) (Fig. 3) . El valor TGS correspondiente n este punto fue de 49.99 u.a. El análisis de regresión logística binaria mostró que los sujetos con un TGS más alto de 49.99 u.a. tenían un OR de 3.310 (95% CI: 2.084-5.528; p < 0.001) de ser atletas profesionales, en comparación con aquellos con un TGS por debajo de este valor. Los deportistas de resistencia de élite mostraron una OR en el punto de corte en comparación con la población no deportista de 4.148 (95% CI: 2.376 7.240; p < 0.001) y los jugadores de fútbol profesional, en comparación con los sujetos no deportistas, tenían una OR de 2.612 (95% CI: 1.519-4.491; p = 0, 001) . La distribución del genotipo de polimorfismos de eficiencia metabólica en el grupo de atletas profesionales en comparación con la población de no atletas fue estadísticamente significativa para el polimorfismo HFE c.187C>G (p = 0.001) , mostrando una mayor frecuencia en el genotipo "óptimo" en atletas (GG 5.80%) que la población no deportista (GG 0.00%) , así como en el genotipo AMPD1 CC (94.20% vs. 62, 10% respectivamente; p = 0.006) y el polimorfismo CYP2D6 c.506-1G>A "óptimo" (p < 0.001) (Tabla 1) . Por otro lado, los datos demostraron que, en el polimorfismo HFE c.187C>G existían diferencias estadísticamente significativas entre ambos grupos de deportistas de resistencia (p < 0.001) y futbolistas profesionales (p = 0.013) con respecto a la población no deportista. En cuanto al polimorfismo AMPD1, también se encontraron diferencias entre deportistas de resistencia, y la población no deportista (p = 0.010) y futbolistas profesionales y no deportistas (p = 0.014) . Igualmente, en el polimorfismo CYP2D6 c.506-1G>A se encontraron diferencias estadísticamente significativas entre ambos grupos de deportistas de resistencia y futbolistas profesionales y la población no deportista (p < 0.001) (tabla 2) . Entre los dos grupos de deportistas profesionales (jugadores de fútbol y de resistencia) , se encontraron resultados estadísticamente significativos en los polimorfismos de HFE c.187C>G mostrando genotipos más favorables en los atletas de resistencia frente a los jugadores de fútbol (p = 0.001) , así como en el polimorfismo GSTP c.313A>G (p = 0.014) , sin embargo en el polimorfismo CYP2D6 c.506-1G>A se presentaron genotipos más favorables en los futbolistas profesionales frente a los atletas de resistencia (p = 0.002) (Tabla 2) . Tabla 1. Distribución de genotipos en atletas profesionales y no atletas de polimorfismos de eficiencia metabolizadora Tabla 2 Para comprobar los perfiles que presentaban un TGS predictivo de eficiencia metabólica entre la población deportista y no deportista, se analizaron combinaciones de 4 en 4 polimorfismos, y de tres en tres polimorfismos. Para ello, las puntuaciones obtenidas en cada polimorfismo genético se sumaron para obtener una puntuación de genotipo total perfecto que representa el genotipo óptimo, y que fue de 8 u.a (rango 0-8 u.a) . Finalmente, este valor se transformó en una escala de 0-100 u.a para facilitar la interpretación mediante la puntuación total del genotipo (TGS) en esta combinación de 4 polimorfismos. Se realizó el mismo cálculo para las combinaciones de tres polimorfismos. 1. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) y GSTT +/- Los resultados de la combinación de estos cuatro polimorfismos mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 64.31 u.a.) frente a la población no deportista (TGS = 55.85 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, igualmente hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 66.83 u.a.) frente a los futbolistas profesionales (TGS = 62.17 u.a.) (p=0.003) , Estos datos sugirieron que, la combinación de estos cuatro polimorfismos tuvo un valor predictivo positivo de eficiencia metabólica y pudiéndose discriminar dentro de las diferentes modalidades deportivas. 2. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) y GSTT +/-y HFE c.187C>G (rs1799945) Los resultados muestran un valor de asociación positiva en los deportistas profesionales (TGS = 60.34 u.a.) frente a la población no deportista (TGS = 54.91 u.a.) (p=0.002) , al igual que dentro de los grupos de los deportistas, donde se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 58.14 u.a.) (p=0.013) , presentando discriminación en el TGS en estos polimorfismos entre las modalidades deportivas. 3. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron en estos polimorfismos un valor de asociación positiva en los deportistas profesionales (TGS = 74.64 u.a.) frente a la población no deportista (TGS = 69.56 u.a.) (p=0.005) , al contrario que lo observado en los grupos de los deportistas, en los que no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 76.33 u.a.) frente a los futbolistas profesionales (TGS = 73.20 u.a.) (p=0.124) , no presentando discriminación en el TGS en estos polimorfismos entre las modalidades deportivas. 4. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y HFE c.187C>G (rs1799945) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.34 u.a.) frente a la población no deportista (TGS = 54.91 u.a.) (p=0.002) . Dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 58.14 u.a.) (p=0.013) , lo que mostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 5. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y HFEc.187C>G (rs1799945) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 69.41 u.a.) frente a la población no deportista (TGS = 62.09 u.a.) (p<0.001) . Igualmente, dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 71.31 u.a.) frente a los futbolistas profesionales (TGS = 67.80 u.a.) (p=0.038) , lo que significa que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos, favoreciendo esta eficiencia metabólica en términos genéticos a los deportistas de resistencia. 6. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) y PGC1ac.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 83.81 u.a.) frente a la población no deportista (TGS = 76.43 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con un TGS de 84.70 u.a. en los deportistas de resistencia frente a un TGS = 83.04 u.a en los futbolistas profesionales (p=0.335) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 7. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTM1 +/-, GSTP1 c.313A>G (rs1695) , CYP2D6 c.506-1G>A (rs3892097) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 71.15 u.a.) frente a la población no deportista (TGS = 62.91 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, se presentaron diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.88 u.a.) frente a los futbolistas (TGS = 68.84 u.a.) (p=0.008) , lo que significa que dentro de las diferentes modalidades deportivas existió discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 8. Polimorfismos AMPD1 c.34C>T (rs17602729) , GSTP1 c.313A>G (rs1695) , GSTT+/-y CYP2D6 c.506-1G>A (rs3892097) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 78.48 u.a.) frente a la población no deportista (TGS = 71.51 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 79.24 u.a.) frente a los futbolistas profesionales (TGS = 77.84 u.a.) (p=0.441) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 9. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/-y GSTP1 c.313A>G (rs1695) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.02 u.a.) frente a la población no deportista (TGS = 46.31 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.58 u.a.) frente a los futbolistas profesionales (TGS = 49.14 u.a.) (p<0.001) , lo que significó que dentro de las diferentes modalidades deportivas existía discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 10. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) , GSTM1 +/- y GSTT +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 50.92 u.a.) frente a la población no deportista (TGS = 42.11 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 54.35 u.a.) frente a los futbolistas (TGS = 48.01 u.a.) (p=0.004) , lo que demostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 11. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y GSTM1 +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.09 u.a.) frente a la población no deportista (TGS = 48.97 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.72 u.a.) frente a los futbolistas profesionales (TGS = 57.85 u.a.) (p=0.006) , lo que demostró que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 12. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y GSTT +/- Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 67.41 u.a.) frente a la población no deportista (TGS = 57.58 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 68.08 u.a.) frente a los futbolistas profesionales (TGS = 66.85 u.a.) (p=0.505) , lo que significó que dentro de las diferentes modalidades deportivas no hubo discriminación en el TGS en estos polimorfismos. 13. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.25 u.a.) frente a la población no deportista (TGS = 47.02 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 59.82 u.a.) frente a los futbolistas profesionales (TGS = 53.21 u.a.) (p=0.001) , lo que significó que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. 14. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , CYP2D6 c.506-1G>A (rs3892097) y PGC1 c.1444 G>A (rs8192678) Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 72.74 u.a.) frente a la población no deportista (TGS = 62.50 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.54 u.a.) frente a los futbolistas profesionales (TGS = 72.06 u.a.) (p=0.351) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 15. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , GSTT+/-y PGC1 c.1444 G>A (rs8192678) . Los resultados muestran un valor de asociación positiva en los deportistas profesionales (TGS = 63.57 u.a.) frente a la población no deportista (TGS = 55.63 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 65.17 u.a.) frente a los futbolistas profesionales (TGS = 62.21 u.a.) (p=0.138) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 16. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , GSTM1 +/- y PGC1 c.1444 G>A (rs8192678) . Los resultados mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.25 u.a.) frente a la población no deportista (TGS = 55.63 u.a.) (p<0.001) . Dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 59.82 u.a.) frente a los futbolistas profesionales (TGS = 53.21 u.a.) (p=0.001) , lo que significó que dentro de las diferentes modalidades deportivas hubo discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia frente a los jugadores de fútbol profesionales. En estas combinaciones de los 4 polimorfismos en la eficiencia metabólica, se mostró discriminación mediante TGS entre deportistas y población no deportista en todas las combinaciones posibles, presentando a su vez dentro de los dos grupos de deportistas el grupo de resistencia una mayor puntuación TFS en los marcadores seleccionados, presentando mejor selección adaptativa en términos genéticos en los polimorfismos implicados en eficiencia metabólica. Se estimó que la combinación más predictiva de deportistas y la discriminación entre el grupo de resistencia y futbolistas profesionales fue la combinación de cuatro polimorfismos, como se ha comprobado anteriormente, debido a que supera el puntaje del 50% del total, lo que otorga validez a esta estimación frente a las combinaciones de menos genes debido a la probabilidad de predecir falsos positivos en el perfil genético de eficiencia metabólica Para ello, se trabajó con diversas combinaciones para mostrar este valor predictivo en los TGS de únicamente 3 polimorfismos diferentes; 17. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , PGC1 c.1444 G>A (rs8192678) . Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 65.40 u.a.) frente a la población no deportista (TGS = 56.55 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 67.28 u.a.) frente a los futbolistas (TGS = 63.85 u.a.) (p=0.069) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 18. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , y CYP2D6 c.506-1G>A (rs3892097) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 60.04 u.a.) frente a la población no deportista (TGS = 41.53 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo iferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.94 u.a.) frente a los futbolistas profesionales (TGS = 57.57 u.a.) (p=0.130) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 19. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , y GSTM1 +/- Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 59.64 u.a.) frente a la población no deportista (TGS = 51.31 u.a.) (p=0.003) . Igualmente, dentro de los grupos de los deportistas, hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 68.39 u.a.) frente a los futbolistas profesionales (TGS = 52.22 u.a.) (p<0.001) , lo que significó que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos, favoreciendo a los deportistas de resistencia. 20. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFEc.187C>G (rs1799945) , y GSTP1 c.313A>G (rs1695) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 71.59 u.a.) frente a la población no deportista (TGS = 68.29 u.a.) (p=0.448) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 73.60 u.a.) frente a los futbolistas profesionales (TGS = 69.89 u.a.) (p=0.398) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 21. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) y GSTT +/- Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.71 u.a.) frente a la población no deportista (TGS = 43.60 u.a.) (p=0.003) . Sin embargo, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.76 u.a.) frente a los futbolistas profesionales (TGS = 50.27 u.a.) (p=0.058) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 22. Polimorfismos AMPD1 c.34C>T (rs17602729) , PGC1a c.1444G>A (rs8192678) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 56.98 u.a.) frente a la población no deportista (TGS = 53.13 u.a.) (p=0.219) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 57.93 u.a.) frente a los futbolistas (TGS = 56.94 u.a.) (p=0.436) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 23. Polimorfismos AMPD1 c.34C>T (rs17602729) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 36.60 u.a.) frente a la población no deportista (TGS = 33.60 u.a.) (p=0.127) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 38.01 u.a.) frente a los futbolistas (TGS = 35.87 u.a.) (p=0.195) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 24. Polimorfismos PGC1a c.1444G>A (rs8192678) , HFE c.187C>G (rs1799945) , HFE c.845G>A (rs1800562) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 36.52 u.a.) frente a la población no deportista (TGS = 30.05 u.a.) (p<0.001) . Al igual que se observó dentro de los grupos de los deportistas, con diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 38.22 u.a.) frente a los futbolistas (TGS = 34.46 u.a.) (p=0.024) , lo que significó que dentro de las diferentes modalidades deportivas hay discriminación en el TGS en estos polimorfismos. 25. Polimorfismos CYP2D6 c.506-1G>A (rs3892097) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 62.95 u.a.) frente a la población no deportista (TGS = 55.73 u.a.) (p<0.001) . Sin embargo, dentro de los grupos de los deportistas, no hubo iferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 62.39 u.a.) frente a los futbolistas (TGS = 63.63 u.a.) (p=0.653) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 26. Polimorfismos GTSM1 +/-, AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 45.34 u.a.) frente a la población no deportista (TGS = 42.13 u.a.) (p=0.098) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 44.62 u.a.) frente a los futbolistas (TGS = 46.21 u.a.) (p=0.350) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 27. Polimorfismos GSTP1 c.313A>G (rs1695) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 53.36 u.a.) frente a la población no deportista (TGS = 50.54 u.a.) (p=0.173) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 54.73 u.a.) frente a los futbolistas (TGS = 52.51 u.a.) (p=0.527) , lo que significa que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 28. Polimorfismos GSTT+/-, AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 49.91 u.a.) frente a la población no deportista (TGS = 46.58 u.a.) (p=0.69) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 49.57 u.a.) frente a los futbolistas (TGS = 50.50 u.a.) (p=0.632) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. 29. Polimorfismos PGC1a c.1444G>A (rs8192678) , AMPD1 c.34C>T (rs17602729) , HFE c.845G>A (rs1800562) Los resultados en esta combinación no mostraron un valor de asociación positiva en los deportistas profesionales (TGS = 55.98 u.a.) frente a la población no deportista (TGS = 53.16 u.a.) (p=0.310) . Igualmente, dentro de los grupos de los deportistas, no hubo diferencias en estos polimorfismos en cuanto al valor predictivo del TFG, con valor en los deportistas de resistencia (TGS = 56.43 u.a.) frente a los futbolistas (TGS = 55.44 u.a.) (p=0.496) , lo que significó que dentro de las diferentes modalidades deportivas no hay discriminación en el TGS en estos polimorfismos. Como se puede apreciar, la combinación de 3 polimorfismos no recoge toda la discriminación en el TGS que sí se observa en todas las combinaciones de 4 polimorfismos entre los deportistas y la población no deportista. Esta combinación de 3 polimorfismos no presenta la capacidad discriminatoria entre grupos debido a la falta de potencia establecida, con una probabilidad alta de predecir falsos positivos en el perfil genético de eficiencia metabólica.
Publicaciones:
ES2958596 (12/02/2024) - A1 Solicitud de patente con informe sobre el estado de la técnica
Eventos:
En fecha 15/07/2022 se realizó Registro Instancia de Solicitud
En fecha 15/07/2022 se realizó Admisión a Trámite
En fecha 15/07/2022 se realizó 1001P_Comunicación Admisión a Trámite
En fecha 28/07/2022 se realizó Superado examen de oficio
En fecha 12/04/2023 se realizó Realizado IET
En fecha 14/04/2023 se realizó 1109P_Comunicación Traslado del IET
En fecha 12/02/2024 se realizó Publicación Solicitud
En fecha 12/02/2024 se realizó Publicación Folleto Solicitud con IET (A1)
En fecha 09/04/2024 se realizó PETEX_Petición de examen sustantivo
Pagos:
15/07/2022 - Pago Tasas IET
Fuente de la información
Parte de la información aquí publicada es pública puesto que ha sido obtenida de la Oficina de Propiedad Industrial de los diferentes países el 27/04/2024 y por lo tanto puede ser que la información no esté actualizada.Parte de la información aquí mostrada ha sido calculada por nuestro sistema informático y puede no ser veraz.
Privacidad
Si considera que al información aquí publicada afecta a su privacidad y desea que eliminemos la información aquí publicada envíe un email a info@patentes-y-marcas.com o rellene el formulario que encontrará aquí.Información sobre el registro de patente nacional por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654
El registro de patente nacional por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654 fue solicitada el 15/07/2022. Se trata de un registro en España por lo que este registro no ofrece protección en el resto de países. El registro MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654 fue solicitada por FUNDACIÓN UNIVERSIDAD FRANCISCO DE VITORIA mediante los servicios del agente Gustavo Adolfo de Dios Serranía. El registro [modality] por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654 está clasificado como C12Q 1/68 según la clasificación internacional de patentes.
Otras invenciones solicitadas por Fundación Universidad Francisco de Vitoria
Es posible conocer todas las invenciones solicitadas por Fundación Universidad Francisco de Vitoria entre las que se encuentra el registro de patente nacional por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654. Si se desean conocer más invenciones solicitadas por Fundación Universidad Francisco de Vitoria clicar aquí.Otras invenciones solicitadas en la clasificación internacional de patentes C12Q 1/68.
Es posible conocer invenciones similares al campo de la técnica se refiere. El registro de patente nacional por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654 está clasificado con la clasificación C12Q 1/68 por lo que si se desea conocer más registros con la clasificación C12Q 1/68 clicar aquí.Otras invenciones solicitadas a través del representante GUSTAVO ADOLFO DE DIOS SERRANÍA
Es posible conocer todas las invenciones solicitadas a través del agente GUSTAVO ADOLFO DE DIOS SERRANÍA entre las que se encuentra el registro patente nacional por MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654. Si se desean conocer más invenciones solicitadas a través del agente GUSTAVO ADOLFO DE DIOS SERRANÍA clicar aquí.Patentes en España
Es posible conocer todas las invenciones publicadas en España entre las que se encuentra el registro patente nacional por MARCADORES DE EFICIENCIA METABÓLICA. Nuestro portal www.patentes-y-marcas.com ofrece acceso a las publicaciones de patentes en España. Conocer las patentes registradas en un país es importante para saber las posibilidades de fabricar, vender o explotar una invención en España.Patentes registradas en la clase C
Es posible conocer todas las patentes registradas en la clase C (QUIMICA; METALURGIA) entre las que se encuentra la patente MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.Patentes registradas en la clase C12
Es posible conocer todas las patentes registradas en la clase C12 (BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUT) entre las que se encuentra la patente MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.Patentes registradas en la clase C12Q
Es posible conocer todas las patentes registradas en la clase C12Q (PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICR) entre las que se encuentra la patente MARCADORES DE EFICIENCIA METABÓLICA con el número P202230654. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.
¿Tienes alguna duda?
Escribe tu consulta y te responderemos rápida y gratuitamente.


 P202230652
P202230652