- Home /
- Publicaciones de patentes /
- MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE
Patente nacional por "MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE"
Este registro ha sido solicitado por
Persona física
a través del representanteMANUEL ILLESCAS TABOADA
Contacto
- Estado: A punto de caducar
- País:
- España
- Fecha solicitud:
- 09/06/2022
- Número solicitud:
-
P202230505
- Número publicación:
-
ES2957491
- Fecha de concesión:
-
- Inventores:
-
Persona física
- Datos del titular:
-
Persona física
- Datos del representante:
-
Manuel Illescas Taboada
- Clasificación Internacional de Patentes:
- C12Q 1/6895
- Clasificación Internacional de Patentes de la publicación:
- C12Q 1/6895
- Fecha de vencimiento:
Quiero registrar una patente
Reivindicaciones:
+ ES-2957491_A11. Un método para identificar una raza hortícola de aguacate que comprende: (a) detectar al menos una región genómica específica de raza hortícola que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, en una muestra de una planta de aguacate; e (b) identificar una raza hortícola de aguacate según las siguientes condiciones: - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a SEQ ID NO: 17 ó SEQ ID NO: 18, se identifica la raza hortícola de la planta de aguacate como Guatemalteca o hídrido de raza Guatemalteca; y - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, se identifica la raza hortícola de la planta de aguacate como Antillana o híbrido de raza Antillana. 2. El método según la reivindicación 1, en el que dicha muestra es una muestra de hojas o de raíces. 3. El método según la reivindicación 1 ó 2, en el que en las etapas (a) y (b) , la identidad de secuencia es del 90%. 4. El método según cualquiera de las reivindicaciones 1 a 3, en el que en las etapas (a) y (b) , la identidad de secuencia es del 100%. 5. El método según cualquiera de las reivindicaciones 1 a 4, en el que en las etapas (a) y (b) , se detecta al menos una región genómica específica de raza hortícola que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 o fragmentos de las mismas. 6. El método según cualquiera de las reivindicaciones 1 a 5, en el que en la etapa (a) , la región genómica se detecta por medio de una técnica seleccionada del grupo que onsiste en: amplificación de la región genómica, hibridación con la región genómica, unión a la región genómica y secuenciación de la región genómica. 7. El método según la reivindicación 6, en el que la amplificación de la región genómica se lleva a cabo mediante una técnica seleccionada del grupo que consiste en: reacción en cadena de la polimerasa (PCR) , amplificación isotérmica, reacción en cadena de la ligasa (LCR) y amplificación de sondas dependiente de ligandos múltiples (MLPA) . 8. El método según la reivindicación 7, en el que PCR es PCR múltiple o PCR en tiempo real. 9. El método según la reivindicación 7, en el que la amplificación isotérmica se selecciona del grupo que consiste en: amplificación isotérmica mediada por bucle (LAMP) , amplificación de recombinasa polimerasa (RPA) , amplificación dependiente de helicasa (HDA) y reacción de amplificación de enzimas de corte (NEAR) . 10. El método según la reivindicación 6, en el que la hibridación con la región genómica utiliza una molécula que hibrida con la región genómica, en el que la molécula comprende una sonda. 11. El método según la reivindicación 10, en el que dicha molécula es un ácido nucleico. 12. El método según cualquiera de las reivindicaciones 1 a 9, en el que la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 13. El método según la reivindicación 11, en el que la identidad de secuencia es del 90%. 14. El método según la reivindicación 11 ó 12, en el que la identidad de secuencia es del 100%. 15. El método según cualquiera de las reivindicaciones 12 a 14, en el que se utiliza cualquier pareja de cebadores que consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 16. Un cebador para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. 17. El cebador según la reivindicación 16, en el que la identidad de secuencia es del 90%. 18. El cebador según la reivindicación 16 ó 17, en el que la identidad de secuencia es del 100%. 19. El cebador según cualquiera de las reivindicaciones 16 a 18, que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. 20. Un kit para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende: - al menos una pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14; y - una enzima seleccionada del grupo que consiste en: una ADN polimerasa, una ADN ligasa, una ADN recombinasa y una ADN helicasa o combinaciones de las mismas. 21. El kit según la reivindicación 20, en el que la enzima es ADN polimerasa. 22. El kit según la reivindicación 20 ó 21, que además comprende una pareja de cebadores que consiste en el par de secuencias SEQ ID NO: 15-16. 23. El kit según cualquiera de las reivindicaciones 20 a 22, en el que la identidad de secuencia es del 90%. 24. El kit según cualquiera de las reivindicaciones 20 a 23, en el que la identidad de secuencia es del 100%. 25. El kit según cualquiera de las reivindicaciones 20 a 24, en el que la pareja de cebadores consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 26. El kit según cualquiera de las reivindicaciones 20 a 25, que además comprende desoxirribonucleótidos trifosfato.
Los productos y servicios protegidos por este registro son:
C12Q 1/6895
Descripciones:
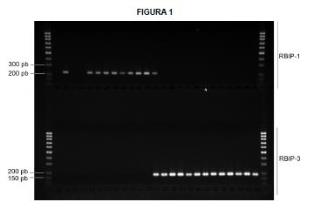
+ ES-2957491_A1 MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE SECTOR DE LA TÉCNICA La presente invención pertenece al sector técnico de la agricultura del aguacate. ANTECEDENTES DE LA INVENCIÓN Existe un gran interés en la identificación por medio de métodos basados en marcadores moleculares de razas hortícolas de aguacate (Persea americana) , dada la importancia creciente de este cultivo. Por parte de la Consejería de Agricultura, Ganadería y Pesca (Gobierno de Canarias) , se recomienda que los ejemplares suministrados por los viveros para su siembra y explotación agronómica sean obtenidos a partir de patrones (parte radicular del ejemplar) , que pertenezcan a la raza hortícola conocida como Antillana (West Indian) . Dicha raza se caracteriza por una mayor resistencia a la salinidad y al hongo fitopatógeno Phytophthora cinnamomi, siendo de especial interés para Canarias. Existe la necesidad de desarrollar un método rápido, escalable, automatizable y con un bajo coste económico, para lo cual se ha desarrollado la presente invención basada en nuevos marcadores moleculares para identificar razas hortícolas de aguacate. DESCRIPCIÓN DE LA INVENCIÓN A efectos de la presente invención, "reacción en cadena de la polimerasa" (PCR, del inglés polymerase chain reaction) se refiere a un método utilizado para hacer rápidamente millones o miles de millones de copias (completas o parciales) de una muestra de ácido desoxirribonucleico (ADN) específica, lo que permite tomar una muestra muy pequeña de ADN y amplificarla (o una parte de ella) a una cantidad lo suficientemente grande como para estudiarla en detalle. A efectos de la presente invención, "PCR en tiempo real" se refiere a una variante de la reacción PCR utilizada para amplificar y simultáneamente hacer un seguimiento en tiempo real del proceso de amplificación de una molécula ADN diana. A efectos de la presente invención, "PCR múltiple" se refiere a una variante de la reacción PCR en la que se amplifican varias secuencias de ADN diferentes imultáneamente. Esta técnica utiliza múltiples parejas de cebadores y una ADN polimerasa. A efectos de la presente invención, "amplificación isotérmica" se refiere a una técnica de amplificación de ADN en la que la reacción se lleva a cabo a una temperatura constante y no requiere un termociclador. A efectos de la presente invención, "reacción en cadena de la ligasa" (LCR, del inglés Ligase Chain Reaction) se refiere a un método de amplificación de ADN en el que se utiliza una ligasa para unir dos sondas u otras moléculas que luego pueden amplificarse mediante reacción PCR u otra técnica de amplificación de ADN. A efectos de la presente invención, "amplificación de sondas dependiente de ligandos múltiples" (MLPA, del inglés Multiplex Ligation-dependent Probe Amplification) se refiere a una variante de la reacción PCR múltiple en el que se detectan cambios en el número de copias a nivel molecular y permite la amplificación de varias moléculas diana con un solo par de cebadores. El problema técnico a resolver consiste en proporcionar un método para identificar razas hortícolas de aguacate basado en la detección de regiones genómicas específicas de raza hortícola de aguacate, así como cebadores y kit para amplificar dichas regiones genómicas específicas. La presente invención soluciona dicho problema técnico. La presente invención proporciona un método para identificar una raza hortícola de aguacate que comprende: (a) detectar al menos una región genómica específica de raza hortícola que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, en una muestra de una planta de aguacate; e (b) identificar una raza hortícola de aguacate según las siguientes condiciones: - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a SEQ ID NO: 17 ó SEQ ID NO: 18, se identifica la raza hortícola de la planta de aguacate como Guatemalteca o hídrido de raza Guatemalteca; y - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, se identifica la raza hortícola de la planta de aguacate como Antillana o híbrido de raza Antillana. Algunas regiones genómicas específicas de raza hortícola de aguacate de la invención contienen una Repetición Terminal Larga (LTR) de un determinado elemento móvil del genoma de la planta de aguacate de tipo retrotransposón, mientras que otras regiones genómicas específicas de raza hortícola de aguacate contienen un locus específico del genoma de la planta de aguacate. Las regiones genómicas con secuencias SEQ ID NO: 17 y SEQ ID NO: 18 son específicas de la raza Guatemalteca y permiten identificar la raza hortícola de aguacate como Guatemalteca o híbrido de la raza Guatemalteca. Las regiones genómicas con secuencias SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 son específicas de la raza Antillana y permiten identificar la raza hortícola de aguacate como Antillana o híbrido de raza Antillana. El método de la invención es rápido, escalable, automatizable y tiene un bajo coste económico. El experto puede determinar si una secuencia tiene al menos un "porcentaje de identidad" determinado respecto a una secuencia de la presente invención, utilizando programas informáticos de alineamiento de secuencias conocidos en el estado de la técnica. Por ejemplo, en la presente memoria se entiende que una secuencia problema tiene al menos un 80% de identidad respecto a una secuencia de referencia de la invención cuando la secuencia puede incluir hasta 20 residuos diferentes por cada 100 residuos de la secuencia de referencia, bien sea por eliminación, inserción o sustitución. Esto se extiende, de forma análoga, al resto de porcentajes de identidad que se describen en la presente memoria. En una realización preferente de la invención, la identidad de secuencia en las etapas (a) y (b) del método de la invención tiene un valor seleccionado del grupo que consiste n: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente del método de la invención, dicha muestra es una muestra de hojas o de raíces. En otra realización preferente del método de la invención, se lleva a cabo una etapa previa de extracción o purificación de ADN genómico a partir de la muestra de la planta de aguacate. En otra realización preferente de la invención, en las etapas (a) y (b) del método de la invención, se detecta al menos una región genómica específica de raza hortícola que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 o fragmentos de las mismas. En otra realización preferente de la invención, en la etapa (a) del método de la invención, la región genómica se detecta por medio de una técnica seleccionada del grupo que consiste en: amplificación de la región genómica, hibridación con la región genómica, unión a la región genómica y secuenciación de la región genómica. En otra realización preferente del método de la invención, la amplificación de la región genómica se lleva a cabo mediante una técnica seleccionada del grupo que consiste en: reacción en cadena de la polimerasa (PCR) , amplificación isotérmica, reacción en cadena de la ligasa (LCR) y amplificación de sondas dependiente de ligandos múltiples (MLPA) . Preferentemente, PCR es PCR múltiple o PCR en tiempo real. Preferentemente, la amplificación isotérmica se selecciona del grupo que consiste en: amplificación isotérmica mediada por bucle (LAMP, del inglés loop-mediated isothermal amplification) , amplificación de recombinasa polimerasa (RPA, del inglés Recombinase Polymerase Amplification) , amplificación dependiente de helicasa (HDA, del inglés Helicase-Dependent Amplification) y reacción de amplificación de enzimas de corte (NEAR, del inglés Nicking Enzyme Amplification Reaction) . En una realización más preferente del método de la invención, se lleva a cabo una etapa final de análisis de resultados mediante electroforesis. Más preferentemente, mediante electroforesis en geles de agarosa. En otra realización preferente del método de la invención, la hibridación con la región genómica utiliza una molécula que hibrida con la región genómica, en el que la molécula comprende una sonda. Preferentemente, dicha molécula es un ácido nucleico. En otra realización preferente del método de la invención, la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. La pareja de cebadores con secuencias SEQ ID NO: 1 y SEQ ID NO: 2 amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 17; la pareja de cebadores con secuencias SEQ ID NO: 3 y SEQ ID NO: 4 amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 18; la pareja de cebadores con secuencias SEQ ID NO: 5 y SEQ ID NO: 6 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 19; la pareja de cebadores con secuencias SEQ ID NO: 7 y SEQ ID NO: 8 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 20; la pareja de cebadores con secuencias SEQ ID NO: 9 y SEQ ID NO: 10 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 21; la pareja de cebadores con secuencias SEQ ID NO: 11 y SEQ ID NO: 12 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 22 y la pareja de cebadores con secuencias SEQ ID NO: 13 y SEQ ID NO: 14 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 23. En una realización más preferente del método de la invención, la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un porcentaje de identidad seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%, respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. En otra realización más preferente del método de la invención, se utiliza cualquier pareja de cebadores que consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. La presente invención también proporciona un cebador para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. En una realización preferente del cebador de la invención, la identidad de secuencia tiene un valor seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente de la invención, el cebador de la invención consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. La presente invención también proporciona un kit para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende: - al menos una pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14; y - una enzima seleccionada del grupo que consiste en: una ADN polimerasa, una ADN ligasa, una ADN recombinasa y una ADN helicasa, o combinaciones de las mismas. En una realización preferente de la invención, la enzima es ADN polimerasa. En otra realización preferente de la invención, el kit de la invención además comprende una pareja de cebadores que consiste en el par de secuencias SEQ ID NO: 15-16. En otra realización preferente del kit de la invención, la identidad de secuencia tiene un valor seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente del kit de la invención, la pareja de cebadores consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. En otra realización preferente de la invención, el kit de la invención además comprende desoxirribonucleótidos trifosfato. En una realización más preferente del kit de la invención, la ADN polimerasa es Taq ADN polimerasa o Phire Hot Star II. En una realización más preferente de la invención, el kit de la invención además comprende reactivos y tampones adecuados para llevar a cabo la amplificación de regiones genómicas. En otra realización más preferente de la invención, el kit de la invención además comprende tampones adecuados para llevar a cabo la amplificación de regiones genómicas. A lo largo de la descripción y las reivindicaciones, el término "comprende", "que comprende" y sus variantes no son de naturaleza limitativa y, por lo tanto, no pretenden excluir otras características técnicas. El término "comprende", "que comprende" y sus variantes, a lo largo de la descripción y las reivindicaciones, incluye, específicamente, el término "consiste en", "que consiste en" y sus variantes. Como se usa en esta descripción y en las reivindicaciones, las formas singulares "el", "la" incluyen referencias a las formas plurales a menos que el contenido indique claramente lo contrario. A menos que se defina lo contrario, todos los términos técnicos y científicos utilizados a lo largo de la descripción y reivindicaciones, tienen el mismo significado que el comúnmente entendido por un experto en el campo de la invención. BREVE DESCRIPCIÓN DE LOS DIBUJOS Figura 1. Análisis de la amplificación por PCR utilizando las parejas de cebadores RBIP-1 y RBIP-3 de 25 cultivares de aguacate. El orden de las muestras es, de izquierda a derecha: Thomas (M) , Puebla (GxM) , Zutano (GxM) , Bacon (GxM) , Fuerte (GxM) , Orotava (GxM) , Pinkerton (GxM) , Hass (GxM) , Lamb-Hass (GxM) , BL667 (GxM) , Arona (G) , Reed (G) , Choquette (GxA) , Julián (GxA) , SS3 (A) , Desconocida procedente de la Gomera (A) , De La Verruga (A) , VGR38 (A) , VGR32 (A) , T23 (A) , BA2 (A) , A3 (A) , Gallo 4 (A) , V1 (A) , M1 (A) . A: Antillano; G: Guatemalteco; M: Mejicano; PM: DNA Ladder 100 bp (AppliChem) . DESCRIPCIÓN DE MODOS DE REALIZACIÓN Ejemplo 1. Identificación de regiones genómicas específicas de raza hortícola de aguacate. Ensayos de amplificación por PCR. Se identificaron regiones genómicas específicas de raza hortícola de aguacate. Las regiones genómicas con secuencias SEQ ID NO: 17 y SEQ ID NO: 18 son específicas de la raza Guatemalteca y permiten identificar la raza hortícola de aguacate como Guatemalteca o híbrido de la raza Guatemalteca (Tabla 1) . Las regiones genómicas con secuencias SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 son específicas de la raza Antillana y permiten identificar la raza hortícola de aguacate como Antillana o híbrido de raza Antillana (Tabla 1) . Se diseñaron las siguientes 7 parejas de cebadores para amplificar por PCR las regiones genómicas específicas de raza hortícola de aguacate identificadas por las secuencias SEQ ID NO: 17-23 (Tabla 1) : • Pareja RBIP-1, con secuencias SEQ ID NO: 1 y SEQ ID NO:2, que amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 17 • Pareja RBIP-2, con secuencias SEQ ID NO: 3 y SEQ ID NO: 4, que amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 18 • Pareja RBIP-3, con secuencias SEQ ID NO: 5 y SEQ ID NO: 6, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 19 • Pareja RBIP-5, con secuencias SEQ ID NO: 7 y SEQ ID NO: 8, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 20 • Pareja RBIP-6, con secuencias SEQ ID NO: 9 y SEQ ID NO: 10, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 21 • Pareja RBIP-C1, con secuencias SEQ ID NO: 11 y SEQ ID NO: 12, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 22 • Pareja RBIP-C2, con secuencias SEQ ID NO: 13 y SEQ ID NO: 14, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 23 Las reacciones PCR se realizaron en un volumen final de 20 l que contienen 5 l de ADN genómico extraído de material vegetal (hojas y raíces) de plantas de aguacate a 0, 4 ng/l (a excepción del control negativo en el que se colocaron 5 l de agua de calidad biología molecular) , 2 l de Key Buffer 10X (contiene MgCh 15 mM) , 2 l de MgCh a 5 mM, 2 l de BSA a 5 g/l, 2 l de cada cebador a 2 M, 0, 1 de l de Taq ADN polimerasa (VWR) y 4, 9 l de agua de calidad biología molecular. Esta enzima, al no poseer actividad de inicio de la reacción en caliente, se añadió a la mezcla maestra de reacción siempre en último lugar, justo después de dispensar las muestras de ADN enómico en los tubos de reacción. Las reacciones se incubaron en un termociclador con el siguiente perfil térmico: desnaturalización inicial a 95°C (2 min) , 35 ciclos de amplificación con desnaturalización a 95°C (10 s) , alineamiento a 60°C (10 s) y extensión a 72°C (20 s) ; extensión final a 72°C (1 min) . Los resultados de las reacciones PCR (5 l de la reacción) se analizaron en geles de agarosa al 2% con 5 cm de recorrido y preparados con tampón TBE 1X. Los geles se corrieron con una diferencia de potencial constante de 80V durante 2 h. Los geles se tiñeron en una solución de bromuro de etidio a 1 g/ml durante 30 min y se fotografiaron en presencia de luz ultravioleta con un sistema de captura de imágenes. Se utilizaron las siguientes parejas de cebadores que amplifican regiones genómicas específicas de raza hortícola de aguacate: RBIP-1, RBIP-2, RBIP-3, RBIP-5, RBIP-6, RBIP-C1 y RBIP-C2, para amplificar 25 cultivares de aguacate diferentes, incluyendo: 11 Antillanos, 2 Guatemaltecos, 1 Mexicano, 9 híbridos Guatemalteco x Mejicano y 2 híbridos Guatemalteco x Antillano. Como control positivo de la amplificación por PCR, se utilizó la pareja de cebadores RBIP-8, que amplificó los 25 cultivares testados. Los resultados de identificación de raza con estas parejas de cebadores que amplifican regiones genómicas específicas de raza hortícola de aguacate se muestran en la Tabla 2. La pareja de cebadores RBIP-3 dio lugar al amplicón esperado (188 pb) en el 100% de los Antillanos testados y los híbridos Guatemalteco x Antillano (Figura 1) y no amplificó en el resto de cultivares, por lo que la región genómica con secuencia SEQ ID NO: 19 es un marcador robusto especifico de la raza Antillana. Con las parejas de cebadores RBIP-1 y RBIP-2, se obtuvieron los amplicones esperados (227 y 283 pb, respectivamente) , regiones genómicas específicas de raza Guatemalteca, en el 75% y 46%, respectivamente, de los cultivares con componente Guatemalteco. Los resultados de amplificación por PCR con la pareja de cebadores RBIP-1 se muestran en la Figura 1. Con las parejas de cebadores RBIP-C1, RBIP-C2, RBIP-5 y RBIP-6, se obtuvieron se obtuvieron los amplicones esperados, regiones genómicas específicas de raza Antillana. Con las parejas de cebadores RBIP-C1 y RBIP-6, se obtuvo el amplicón esperado en un 84, 7% de los cultivares con componente Antillano ensayados. Con las parejas de cebadores RBIP-C2 y RBIP-5, se obtuvo el amplicón esperado en un 77% y 61, 5% de los casos, respectivamente. TEXTO LIBRE DE LA LISTA DE SECUENCIAS El texto libre del listado de secuencias se reproduce en la Tabla 3.
Publicaciones:
ES2957491 (19/01/2024) - A1 Solicitud de patente con informe sobre el estado de la técnica
Eventos:
En fecha 09/06/2022 se realizó Registro Instancia de Solicitud
En fecha 09/06/2022 se realizó Admisión a Trámite
En fecha 09/06/2022 se realizó 1001P_Comunicación Admisión a Trámite
En fecha 23/06/2022 se realizó Superado examen de oficio
En fecha 04/05/2023 se realizó Realizado IET
En fecha 05/05/2023 se realizó 1109P_Comunicación Traslado del IET
En fecha 12/12/2023 se realizó 3411X_Alta Mandatarios
En fecha 12/12/2023 se realizó Alta mandatario
En fecha 29/12/2023 se realizó PETEX_Petición de examen sustantivo
En fecha 19/01/2024 se realizó Publicación Solicitud
En fecha 19/01/2024 se realizó Publicación Folleto Solicitud con IET (A1)
En fecha 06/05/2024 se realizó Validación petición y/o pago de examen sustantivo conforme
Pagos:
08/06/2022 - Pago Tasas IET
+ ES-2957491_A11. Un método para identificar una raza hortícola de aguacate que comprende: (a) detectar al menos una región genómica específica de raza hortícola que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, en una muestra de una planta de aguacate; e (b) identificar una raza hortícola de aguacate según las siguientes condiciones: - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a SEQ ID NO: 17 ó SEQ ID NO: 18, se identifica la raza hortícola de la planta de aguacate como Guatemalteca o hídrido de raza Guatemalteca; y - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, se identifica la raza hortícola de la planta de aguacate como Antillana o híbrido de raza Antillana. 2. El método según la reivindicación 1, en el que dicha muestra es una muestra de hojas o de raíces. 3. El método según la reivindicación 1 ó 2, en el que en las etapas (a) y (b) , la identidad de secuencia es del 90%. 4. El método según cualquiera de las reivindicaciones 1 a 3, en el que en las etapas (a) y (b) , la identidad de secuencia es del 100%. 5. El método según cualquiera de las reivindicaciones 1 a 4, en el que en las etapas (a) y (b) , se detecta al menos una región genómica específica de raza hortícola que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 o fragmentos de las mismas. 6. El método según cualquiera de las reivindicaciones 1 a 5, en el que en la etapa (a) , la región genómica se detecta por medio de una técnica seleccionada del grupo que onsiste en: amplificación de la región genómica, hibridación con la región genómica, unión a la región genómica y secuenciación de la región genómica. 7. El método según la reivindicación 6, en el que la amplificación de la región genómica se lleva a cabo mediante una técnica seleccionada del grupo que consiste en: reacción en cadena de la polimerasa (PCR) , amplificación isotérmica, reacción en cadena de la ligasa (LCR) y amplificación de sondas dependiente de ligandos múltiples (MLPA) . 8. El método según la reivindicación 7, en el que PCR es PCR múltiple o PCR en tiempo real. 9. El método según la reivindicación 7, en el que la amplificación isotérmica se selecciona del grupo que consiste en: amplificación isotérmica mediada por bucle (LAMP) , amplificación de recombinasa polimerasa (RPA) , amplificación dependiente de helicasa (HDA) y reacción de amplificación de enzimas de corte (NEAR) . 10. El método según la reivindicación 6, en el que la hibridación con la región genómica utiliza una molécula que hibrida con la región genómica, en el que la molécula comprende una sonda. 11. El método según la reivindicación 10, en el que dicha molécula es un ácido nucleico. 12. El método según cualquiera de las reivindicaciones 1 a 9, en el que la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 13. El método según la reivindicación 11, en el que la identidad de secuencia es del 90%. 14. El método según la reivindicación 11 ó 12, en el que la identidad de secuencia es del 100%. 15. El método según cualquiera de las reivindicaciones 12 a 14, en el que se utiliza cualquier pareja de cebadores que consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 16. Un cebador para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. 17. El cebador según la reivindicación 16, en el que la identidad de secuencia es del 90%. 18. El cebador según la reivindicación 16 ó 17, en el que la identidad de secuencia es del 100%. 19. El cebador según cualquiera de las reivindicaciones 16 a 18, que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. 20. Un kit para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende: - al menos una pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14; y - una enzima seleccionada del grupo que consiste en: una ADN polimerasa, una ADN ligasa, una ADN recombinasa y una ADN helicasa o combinaciones de las mismas. 21. El kit según la reivindicación 20, en el que la enzima es ADN polimerasa. 22. El kit según la reivindicación 20 ó 21, que además comprende una pareja de cebadores que consiste en el par de secuencias SEQ ID NO: 15-16. 23. El kit según cualquiera de las reivindicaciones 20 a 22, en el que la identidad de secuencia es del 90%. 24. El kit según cualquiera de las reivindicaciones 20 a 23, en el que la identidad de secuencia es del 100%. 25. El kit según cualquiera de las reivindicaciones 20 a 24, en el que la pareja de cebadores consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. 26. El kit según cualquiera de las reivindicaciones 20 a 25, que además comprende desoxirribonucleótidos trifosfato.
Los productos y servicios protegidos por este registro son:
C12Q 1/6895
Descripciones:
+ ES-2957491_A1 MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE SECTOR DE LA TÉCNICA La presente invención pertenece al sector técnico de la agricultura del aguacate. ANTECEDENTES DE LA INVENCIÓN Existe un gran interés en la identificación por medio de métodos basados en marcadores moleculares de razas hortícolas de aguacate (Persea americana) , dada la importancia creciente de este cultivo. Por parte de la Consejería de Agricultura, Ganadería y Pesca (Gobierno de Canarias) , se recomienda que los ejemplares suministrados por los viveros para su siembra y explotación agronómica sean obtenidos a partir de patrones (parte radicular del ejemplar) , que pertenezcan a la raza hortícola conocida como Antillana (West Indian) . Dicha raza se caracteriza por una mayor resistencia a la salinidad y al hongo fitopatógeno Phytophthora cinnamomi, siendo de especial interés para Canarias. Existe la necesidad de desarrollar un método rápido, escalable, automatizable y con un bajo coste económico, para lo cual se ha desarrollado la presente invención basada en nuevos marcadores moleculares para identificar razas hortícolas de aguacate. DESCRIPCIÓN DE LA INVENCIÓN A efectos de la presente invención, "reacción en cadena de la polimerasa" (PCR, del inglés polymerase chain reaction) se refiere a un método utilizado para hacer rápidamente millones o miles de millones de copias (completas o parciales) de una muestra de ácido desoxirribonucleico (ADN) específica, lo que permite tomar una muestra muy pequeña de ADN y amplificarla (o una parte de ella) a una cantidad lo suficientemente grande como para estudiarla en detalle. A efectos de la presente invención, "PCR en tiempo real" se refiere a una variante de la reacción PCR utilizada para amplificar y simultáneamente hacer un seguimiento en tiempo real del proceso de amplificación de una molécula ADN diana. A efectos de la presente invención, "PCR múltiple" se refiere a una variante de la reacción PCR en la que se amplifican varias secuencias de ADN diferentes imultáneamente. Esta técnica utiliza múltiples parejas de cebadores y una ADN polimerasa. A efectos de la presente invención, "amplificación isotérmica" se refiere a una técnica de amplificación de ADN en la que la reacción se lleva a cabo a una temperatura constante y no requiere un termociclador. A efectos de la presente invención, "reacción en cadena de la ligasa" (LCR, del inglés Ligase Chain Reaction) se refiere a un método de amplificación de ADN en el que se utiliza una ligasa para unir dos sondas u otras moléculas que luego pueden amplificarse mediante reacción PCR u otra técnica de amplificación de ADN. A efectos de la presente invención, "amplificación de sondas dependiente de ligandos múltiples" (MLPA, del inglés Multiplex Ligation-dependent Probe Amplification) se refiere a una variante de la reacción PCR múltiple en el que se detectan cambios en el número de copias a nivel molecular y permite la amplificación de varias moléculas diana con un solo par de cebadores. El problema técnico a resolver consiste en proporcionar un método para identificar razas hortícolas de aguacate basado en la detección de regiones genómicas específicas de raza hortícola de aguacate, así como cebadores y kit para amplificar dichas regiones genómicas específicas. La presente invención soluciona dicho problema técnico. La presente invención proporciona un método para identificar una raza hortícola de aguacate que comprende: (a) detectar al menos una región genómica específica de raza hortícola que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, en una muestra de una planta de aguacate; e (b) identificar una raza hortícola de aguacate según las siguientes condiciones: - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a SEQ ID NO: 17 ó SEQ ID NO: 18, se identifica la raza hortícola de la planta de aguacate como Guatemalteca o hídrido de raza Guatemalteca; y - si se detecta una región genómica que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23, se identifica la raza hortícola de la planta de aguacate como Antillana o híbrido de raza Antillana. Algunas regiones genómicas específicas de raza hortícola de aguacate de la invención contienen una Repetición Terminal Larga (LTR) de un determinado elemento móvil del genoma de la planta de aguacate de tipo retrotransposón, mientras que otras regiones genómicas específicas de raza hortícola de aguacate contienen un locus específico del genoma de la planta de aguacate. Las regiones genómicas con secuencias SEQ ID NO: 17 y SEQ ID NO: 18 son específicas de la raza Guatemalteca y permiten identificar la raza hortícola de aguacate como Guatemalteca o híbrido de la raza Guatemalteca. Las regiones genómicas con secuencias SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 son específicas de la raza Antillana y permiten identificar la raza hortícola de aguacate como Antillana o híbrido de raza Antillana. El método de la invención es rápido, escalable, automatizable y tiene un bajo coste económico. El experto puede determinar si una secuencia tiene al menos un "porcentaje de identidad" determinado respecto a una secuencia de la presente invención, utilizando programas informáticos de alineamiento de secuencias conocidos en el estado de la técnica. Por ejemplo, en la presente memoria se entiende que una secuencia problema tiene al menos un 80% de identidad respecto a una secuencia de referencia de la invención cuando la secuencia puede incluir hasta 20 residuos diferentes por cada 100 residuos de la secuencia de referencia, bien sea por eliminación, inserción o sustitución. Esto se extiende, de forma análoga, al resto de porcentajes de identidad que se describen en la presente memoria. En una realización preferente de la invención, la identidad de secuencia en las etapas (a) y (b) del método de la invención tiene un valor seleccionado del grupo que consiste n: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente del método de la invención, dicha muestra es una muestra de hojas o de raíces. En otra realización preferente del método de la invención, se lleva a cabo una etapa previa de extracción o purificación de ADN genómico a partir de la muestra de la planta de aguacate. En otra realización preferente de la invención, en las etapas (a) y (b) del método de la invención, se detecta al menos una región genómica específica de raza hortícola que consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 o fragmentos de las mismas. En otra realización preferente de la invención, en la etapa (a) del método de la invención, la región genómica se detecta por medio de una técnica seleccionada del grupo que consiste en: amplificación de la región genómica, hibridación con la región genómica, unión a la región genómica y secuenciación de la región genómica. En otra realización preferente del método de la invención, la amplificación de la región genómica se lleva a cabo mediante una técnica seleccionada del grupo que consiste en: reacción en cadena de la polimerasa (PCR) , amplificación isotérmica, reacción en cadena de la ligasa (LCR) y amplificación de sondas dependiente de ligandos múltiples (MLPA) . Preferentemente, PCR es PCR múltiple o PCR en tiempo real. Preferentemente, la amplificación isotérmica se selecciona del grupo que consiste en: amplificación isotérmica mediada por bucle (LAMP, del inglés loop-mediated isothermal amplification) , amplificación de recombinasa polimerasa (RPA, del inglés Recombinase Polymerase Amplification) , amplificación dependiente de helicasa (HDA, del inglés Helicase-Dependent Amplification) y reacción de amplificación de enzimas de corte (NEAR, del inglés Nicking Enzyme Amplification Reaction) . En una realización más preferente del método de la invención, se lleva a cabo una etapa final de análisis de resultados mediante electroforesis. Más preferentemente, mediante electroforesis en geles de agarosa. En otra realización preferente del método de la invención, la hibridación con la región genómica utiliza una molécula que hibrida con la región genómica, en el que la molécula comprende una sonda. Preferentemente, dicha molécula es un ácido nucleico. En otra realización preferente del método de la invención, la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. La pareja de cebadores con secuencias SEQ ID NO: 1 y SEQ ID NO: 2 amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 17; la pareja de cebadores con secuencias SEQ ID NO: 3 y SEQ ID NO: 4 amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 18; la pareja de cebadores con secuencias SEQ ID NO: 5 y SEQ ID NO: 6 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 19; la pareja de cebadores con secuencias SEQ ID NO: 7 y SEQ ID NO: 8 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 20; la pareja de cebadores con secuencias SEQ ID NO: 9 y SEQ ID NO: 10 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 21; la pareja de cebadores con secuencias SEQ ID NO: 11 y SEQ ID NO: 12 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 22 y la pareja de cebadores con secuencias SEQ ID NO: 13 y SEQ ID NO: 14 amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 23. En una realización más preferente del método de la invención, la amplificación de la región genómica utiliza cualquier pareja de cebadores que comprende secuencias con al menos un porcentaje de identidad seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%, respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. En otra realización más preferente del método de la invención, se utiliza cualquier pareja de cebadores que consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. La presente invención también proporciona un cebador para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende una secuencia con al menos un 80% de identidad respecto a cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. En una realización preferente del cebador de la invención, la identidad de secuencia tiene un valor seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente de la invención, el cebador de la invención consiste en cualquiera de las secuencias seleccionadas del grupo que consiste en: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 y SEQ ID NO: 14. La presente invención también proporciona un kit para amplificar regiones genómicas específicas de raza hortícola de aguacate que comprende: - al menos una pareja de cebadores que comprende secuencias con al menos un 80% de identidad respecto a cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14; y - una enzima seleccionada del grupo que consiste en: una ADN polimerasa, una ADN ligasa, una ADN recombinasa y una ADN helicasa, o combinaciones de las mismas. En una realización preferente de la invención, la enzima es ADN polimerasa. En otra realización preferente de la invención, el kit de la invención además comprende una pareja de cebadores que consiste en el par de secuencias SEQ ID NO: 15-16. En otra realización preferente del kit de la invención, la identidad de secuencia tiene un valor seleccionado del grupo que consiste en: 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y 100%. En otra realización preferente del kit de la invención, la pareja de cebadores consiste en cualquiera de los pares de secuencias seleccionados del grupo que consiste en: SEQ ID NO: 1-2, SEQ ID NO: 3-4, SEQ ID NO: 5-6, SEQ ID NO: 7-8, SEQ ID NO: 9-10, SEQ ID NO: 11-12 y SEQ ID NO: 13-14. En otra realización preferente de la invención, el kit de la invención además comprende desoxirribonucleótidos trifosfato. En una realización más preferente del kit de la invención, la ADN polimerasa es Taq ADN polimerasa o Phire Hot Star II. En una realización más preferente de la invención, el kit de la invención además comprende reactivos y tampones adecuados para llevar a cabo la amplificación de regiones genómicas. En otra realización más preferente de la invención, el kit de la invención además comprende tampones adecuados para llevar a cabo la amplificación de regiones genómicas. A lo largo de la descripción y las reivindicaciones, el término "comprende", "que comprende" y sus variantes no son de naturaleza limitativa y, por lo tanto, no pretenden excluir otras características técnicas. El término "comprende", "que comprende" y sus variantes, a lo largo de la descripción y las reivindicaciones, incluye, específicamente, el término "consiste en", "que consiste en" y sus variantes. Como se usa en esta descripción y en las reivindicaciones, las formas singulares "el", "la" incluyen referencias a las formas plurales a menos que el contenido indique claramente lo contrario. A menos que se defina lo contrario, todos los términos técnicos y científicos utilizados a lo largo de la descripción y reivindicaciones, tienen el mismo significado que el comúnmente entendido por un experto en el campo de la invención. BREVE DESCRIPCIÓN DE LOS DIBUJOS Figura 1. Análisis de la amplificación por PCR utilizando las parejas de cebadores RBIP-1 y RBIP-3 de 25 cultivares de aguacate. El orden de las muestras es, de izquierda a derecha: Thomas (M) , Puebla (GxM) , Zutano (GxM) , Bacon (GxM) , Fuerte (GxM) , Orotava (GxM) , Pinkerton (GxM) , Hass (GxM) , Lamb-Hass (GxM) , BL667 (GxM) , Arona (G) , Reed (G) , Choquette (GxA) , Julián (GxA) , SS3 (A) , Desconocida procedente de la Gomera (A) , De La Verruga (A) , VGR38 (A) , VGR32 (A) , T23 (A) , BA2 (A) , A3 (A) , Gallo 4 (A) , V1 (A) , M1 (A) . A: Antillano; G: Guatemalteco; M: Mejicano; PM: DNA Ladder 100 bp (AppliChem) . DESCRIPCIÓN DE MODOS DE REALIZACIÓN Ejemplo 1. Identificación de regiones genómicas específicas de raza hortícola de aguacate. Ensayos de amplificación por PCR. Se identificaron regiones genómicas específicas de raza hortícola de aguacate. Las regiones genómicas con secuencias SEQ ID NO: 17 y SEQ ID NO: 18 son específicas de la raza Guatemalteca y permiten identificar la raza hortícola de aguacate como Guatemalteca o híbrido de la raza Guatemalteca (Tabla 1) . Las regiones genómicas con secuencias SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22 y SEQ ID NO: 23 son específicas de la raza Antillana y permiten identificar la raza hortícola de aguacate como Antillana o híbrido de raza Antillana (Tabla 1) . Se diseñaron las siguientes 7 parejas de cebadores para amplificar por PCR las regiones genómicas específicas de raza hortícola de aguacate identificadas por las secuencias SEQ ID NO: 17-23 (Tabla 1) : • Pareja RBIP-1, con secuencias SEQ ID NO: 1 y SEQ ID NO:2, que amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 17 • Pareja RBIP-2, con secuencias SEQ ID NO: 3 y SEQ ID NO: 4, que amplifica la región genómica específica de raza Guatemalteca con secuencia SEQ ID NO: 18 • Pareja RBIP-3, con secuencias SEQ ID NO: 5 y SEQ ID NO: 6, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 19 • Pareja RBIP-5, con secuencias SEQ ID NO: 7 y SEQ ID NO: 8, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 20 • Pareja RBIP-6, con secuencias SEQ ID NO: 9 y SEQ ID NO: 10, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 21 • Pareja RBIP-C1, con secuencias SEQ ID NO: 11 y SEQ ID NO: 12, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 22 • Pareja RBIP-C2, con secuencias SEQ ID NO: 13 y SEQ ID NO: 14, que amplifica la región genómica específica de raza Antillana con secuencia SEQ ID NO: 23 Las reacciones PCR se realizaron en un volumen final de 20 l que contienen 5 l de ADN genómico extraído de material vegetal (hojas y raíces) de plantas de aguacate a 0, 4 ng/l (a excepción del control negativo en el que se colocaron 5 l de agua de calidad biología molecular) , 2 l de Key Buffer 10X (contiene MgCh 15 mM) , 2 l de MgCh a 5 mM, 2 l de BSA a 5 g/l, 2 l de cada cebador a 2 M, 0, 1 de l de Taq ADN polimerasa (VWR) y 4, 9 l de agua de calidad biología molecular. Esta enzima, al no poseer actividad de inicio de la reacción en caliente, se añadió a la mezcla maestra de reacción siempre en último lugar, justo después de dispensar las muestras de ADN enómico en los tubos de reacción. Las reacciones se incubaron en un termociclador con el siguiente perfil térmico: desnaturalización inicial a 95°C (2 min) , 35 ciclos de amplificación con desnaturalización a 95°C (10 s) , alineamiento a 60°C (10 s) y extensión a 72°C (20 s) ; extensión final a 72°C (1 min) . Los resultados de las reacciones PCR (5 l de la reacción) se analizaron en geles de agarosa al 2% con 5 cm de recorrido y preparados con tampón TBE 1X. Los geles se corrieron con una diferencia de potencial constante de 80V durante 2 h. Los geles se tiñeron en una solución de bromuro de etidio a 1 g/ml durante 30 min y se fotografiaron en presencia de luz ultravioleta con un sistema de captura de imágenes. Se utilizaron las siguientes parejas de cebadores que amplifican regiones genómicas específicas de raza hortícola de aguacate: RBIP-1, RBIP-2, RBIP-3, RBIP-5, RBIP-6, RBIP-C1 y RBIP-C2, para amplificar 25 cultivares de aguacate diferentes, incluyendo: 11 Antillanos, 2 Guatemaltecos, 1 Mexicano, 9 híbridos Guatemalteco x Mejicano y 2 híbridos Guatemalteco x Antillano. Como control positivo de la amplificación por PCR, se utilizó la pareja de cebadores RBIP-8, que amplificó los 25 cultivares testados. Los resultados de identificación de raza con estas parejas de cebadores que amplifican regiones genómicas específicas de raza hortícola de aguacate se muestran en la Tabla 2. La pareja de cebadores RBIP-3 dio lugar al amplicón esperado (188 pb) en el 100% de los Antillanos testados y los híbridos Guatemalteco x Antillano (Figura 1) y no amplificó en el resto de cultivares, por lo que la región genómica con secuencia SEQ ID NO: 19 es un marcador robusto especifico de la raza Antillana. Con las parejas de cebadores RBIP-1 y RBIP-2, se obtuvieron los amplicones esperados (227 y 283 pb, respectivamente) , regiones genómicas específicas de raza Guatemalteca, en el 75% y 46%, respectivamente, de los cultivares con componente Guatemalteco. Los resultados de amplificación por PCR con la pareja de cebadores RBIP-1 se muestran en la Figura 1. Con las parejas de cebadores RBIP-C1, RBIP-C2, RBIP-5 y RBIP-6, se obtuvieron se obtuvieron los amplicones esperados, regiones genómicas específicas de raza Antillana. Con las parejas de cebadores RBIP-C1 y RBIP-6, se obtuvo el amplicón esperado en un 84, 7% de los cultivares con componente Antillano ensayados. Con las parejas de cebadores RBIP-C2 y RBIP-5, se obtuvo el amplicón esperado en un 77% y 61, 5% de los casos, respectivamente. TEXTO LIBRE DE LA LISTA DE SECUENCIAS El texto libre del listado de secuencias se reproduce en la Tabla 3.
Publicaciones:
ES2957491 (19/01/2024) - A1 Solicitud de patente con informe sobre el estado de la técnica
Eventos:
En fecha 09/06/2022 se realizó Registro Instancia de Solicitud
En fecha 09/06/2022 se realizó Admisión a Trámite
En fecha 09/06/2022 se realizó 1001P_Comunicación Admisión a Trámite
En fecha 23/06/2022 se realizó Superado examen de oficio
En fecha 04/05/2023 se realizó Realizado IET
En fecha 05/05/2023 se realizó 1109P_Comunicación Traslado del IET
En fecha 12/12/2023 se realizó 3411X_Alta Mandatarios
En fecha 12/12/2023 se realizó Alta mandatario
En fecha 29/12/2023 se realizó PETEX_Petición de examen sustantivo
En fecha 19/01/2024 se realizó Publicación Solicitud
En fecha 19/01/2024 se realizó Publicación Folleto Solicitud con IET (A1)
En fecha 06/05/2024 se realizó Validación petición y/o pago de examen sustantivo conforme
Pagos:
08/06/2022 - Pago Tasas IET
Fuente de la información
Parte de la información aquí publicada es pública puesto que ha sido obtenida de la Oficina de Propiedad Industrial de los diferentes países el 09/05/2024 y por lo tanto puede ser que la información no esté actualizada.Parte de la información aquí mostrada ha sido calculada por nuestro sistema informático y puede no ser veraz.
Privacidad
Si considera que al información aquí publicada afecta a su privacidad y desea que eliminemos la información aquí publicada envíe un email a info@patentes-y-marcas.com o rellene el formulario que encontrará aquí.Información sobre el registro de patente nacional por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505
El registro de patente nacional por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505 fue solicitada el 09/06/2022. Se trata de un registro en España por lo que este registro no ofrece protección en el resto de países. El registro MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505 fue solicitada por UNIVERSIDAD DE LA LAGUNA mediante los servicios del agente Manuel Illescas Taboada. El registro [modality] por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505 está clasificado como C12Q 1/6895 según la clasificación internacional de patentes.
Otras invenciones solicitadas en la clasificación internacional de patentes C12Q 1/6895.
Es posible conocer invenciones similares al campo de la técnica se refiere. El registro de patente nacional por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505 está clasificado con la clasificación C12Q 1/6895 por lo que si se desea conocer más registros con la clasificación C12Q 1/6895 clicar aquí.Otras invenciones solicitadas a través del representante MANUEL ILLESCAS TABOADA
Es posible conocer todas las invenciones solicitadas a través del agente MANUEL ILLESCAS TABOADA entre las que se encuentra el registro patente nacional por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505. Si se desean conocer más invenciones solicitadas a través del agente MANUEL ILLESCAS TABOADA clicar aquí.Patentes en España
Es posible conocer todas las invenciones publicadas en España entre las que se encuentra el registro patente nacional por MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE. Nuestro portal www.patentes-y-marcas.com ofrece acceso a las publicaciones de patentes en España. Conocer las patentes registradas en un país es importante para saber las posibilidades de fabricar, vender o explotar una invención en España.Patentes registradas en la clase C
Es posible conocer todas las patentes registradas en la clase C (QUIMICA; METALURGIA) entre las que se encuentra la patente MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.Patentes registradas en la clase C12
Es posible conocer todas las patentes registradas en la clase C12 (BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUT) entre las que se encuentra la patente MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.Patentes registradas en la clase C12Q
Es posible conocer todas las patentes registradas en la clase C12Q (PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICR) entre las que se encuentra la patente MÉTODO, CEBADORES Y KIT PARA IDENTIFICAR REGIONES GENÓMICAS ESPECÍFICAS DE RAZA HORTÍCOLA DE AGUACATE con el número P202230505. Conocer las patentes registradas en una clase es importante para saber las posibilidades de registrar una patente en esa misma clase.
¿Tienes alguna duda?
Escribe tu consulta y te responderemos rápida y gratuitamente.

Otras patentes similares

 P202230503
P202230503